|
|
Las proteínas asociadas al ADN se conocen colectivamente con el nombre de histonas. Son polipéptidos relativamente cortos cargados positivamente (básicos) y por lo tanto son atraídos por las cargas negativas del ADN (ácido) Las histonas son sintetizadas en cantidad durante la fase S ( S por síntesis) del ciclo celular. Una de las funciones de esas proteínas está relacionada con el empaquetamiento del ADN en la forma del cromosoma: los 2 metros de ADN de la célula humana son empaquetados en 46 cromosomas de un largo combinado de aproximadamente 200 nm. La célula tiene unas 90 millones de moléculas de histonas siendo la mayoría perteneciente a un tipo conocido como H1. Se conocen cinco tipos de las siguientes histonas (H1, H2A, H2B, H3, y H4 , 8 moléculas en total); con la excepción de la H1 la mayor parte de las histonas de los eucariotas son muy similares.
Imagen modificada de la University of Illinois' DNA and Protein Synthesis site. El nucleosoma es la unidad fundamental de "empaquetamiento" del ADN eucariótico. El "carretel" ("core") del mismo consiste en dos moléculas de H2A, H2B, H3, y H4; alrededor de las cuales el ADN se enrolla dos veces (1). La histona 1 esta fuera del "carretel". Este nivel de empaquetamiento ("packing") se conoce como "cuentas de un collar" (2). El siguiente nivel se conoce como la fibra de 30 nm, cuyos detalles de organización no se conocen completamente. Las fibras se condensa a posteriori en dominios en bucle de 300 nm (3). Los dominios son parte de las secciones condensadas (4) ( 700 nm) de los cromosomas (el cromosoma tiene un ancho de unos 1.400 nm en la metafase) (5).
Replicación
del cromosoma de los eucariotas
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Existen secuencias altamente repetitivas (de función desconocida, salvo para la secuenciación génica del hombre):
| |||||||
|
Secuencias moderadamente repetitivas:
|
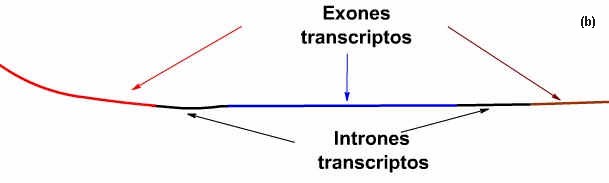
Las secuencias que codifican proteínas están interrumpidas por regiones que no codifican. Las secuencias no codificantes se denominan intrones. Las secuencias codificantes exones.
 |
 |
|
|
| Transcripción, eliminación de los segmentos resultantes de los intrones transcriptos, y unión de los exones. Escenas seleccionadas de Animación de Intrones y exones |
La mayor parte de los genes estructurales de los eucariotas contienen intrones. Si bien se transcriben, estos intrones son cortados (solo en apariencia un proceso ineficiente) antes de la síntesis proteica. Es decir el ARN sufre un proceso de edición, que puede resultar en diferentes péptidos (para el caso de un mARN). El número de intrones varía para cada gen, pueden encontrarse inclusive en tARN y genes virales!. Generalmente cuanto mas complejo y de reciente evolución el organismo, mas numerosos y grandes los intrones. ¿Que surgió primero? ¿genes continuos sin intrones o genes interrumpidos por intrones?. Se ha propuesto que los intrones promueven la recombinación genética (vía crossing-over), y por lo tanto aumentan la velocidad de evolución. Los exones codifican diferentes regiones funcionales de las proteínas.

Imagen modificada de McGill University's Genetics Pages (http://www.mcgill.ca/nrs/regenk_i.htm).
Las
familias de genes
están formadas de genes similares pero no idénticos.
La familia de genes de la globina es la familia de genes mejor estudiada.
La hemoglobina consiste (en el caso humano) en dos cadenas a y dos cadenas
b alrededor de un grupo Hemo. Los genes de la beta globina se encuentran
distribuidos en cinco loci en el cromosoma humano Nro. 11. Estos genes
se expresan secuencialmente durante el desarrollo y tienen intrones de
la misma longitud en posiciones similares en cada gen.
Algunos de estos genes son copias inactivas, otros solo son funcionales
durante determinada fase del desarrollo (recordar la hemoglobina fetal
y la adulta). La familia de genes de la actina, tiene un patrón
similar.
El proceso de transcripción en los eucariotas es similar a los de
los procariotas, existen sin embargo algunas diferencias. Los genes eucariotas
no se agrupan en operones como los de los procariotas. Cada gen eucariota
se transcribe separadamente, con un control transcripcional independiente
para cada gen. Si bien los procariotas tienen un solo tipo de ARN
polimerasa para todos los tipos de ARN, los eucariotas tienen una para
cada tipo. Una para el mARN, una para los rARN largos y una tercera para
los rARN cortos y los tARN.
En procariotas la traducción comienza inclusive antes que la
transcripción haya terminado, mientras que en eucariotas tenemos
dos procesos separados en tiempo y localización (recordar la existencia
de una envoltura nuclear). Luego que en el núcleo de la célula
eucariota se transcribe un ARN, el ARN transcripto es extensamente modificado antes de ser exportado al citoplasma. Se
le agrega 7-metilguanina (una base inusual) al extremo 5' del mARN; y
esto resulta esencial para el pegado del mARN al ribosoma. Una ristra de
adeninas (tanto como 200 nucleótidos conocido como poli-A) se agrega
al extremo 3' del mARN luego de la transcripción. La función
de esta "cola" de poli A no se conoce, pero puede usarse para capturar
mARN para estudios. Los intrones se cortan y los exones se colocan juntos
antes que el mARN deje el núcleo
Existen muchos ejemplos de mensajes idénticos procesados por
diferentes métodos, a veces los intrones se tornan exones y viceversa.
Moléculas de proteínas se pegan al mARN y luego se exportan
del núcleo formando partículas llamadas ribonucleoproteínas
(mRNPs) que parecen ayudar en el transporte por los poros nucleares y también en el pegado a los ribosomas.
Hoy sabemos que que existen muchos "genes" que son claramente funcionales (tal como los que sintetizan los siARN o las ribo-llaves), a pesar que no codifican para ninguna proteína y producen únicamente ARN. Claes Wahlestedt, del Instituto Karoliska (Estocolmo, Suecia), dice respecto al término "gen", tendemos a no utilizar el término gen, nos referimos a segmentos que son transcriptos a ARN como una "unidad transcripcional".
Tema ampliado: The Unseen Genome: Gems among the Junk; November 2003; by W. Wayt Gibbs
Mecanismo de interferencia del siARN
![]()
Los
siARNs (del inglés: short interfering RNA, ARN corto de interferencia) son componentes de una gran respuesta antiviral
denominada interferencia
del ARN, una defensa celular descubierta en plantas y gusanos.
Ciertos
virus están compuestos por ARN de doble cadena y la presencia de tan obvio
ARN ajeno a la célula desencadena, en plantas e invertebrados, una
respuesta liderada por la enzima "DICER" ("cubeteadora"?),
que corta el ARN invasor en pequeños pedazos. Otras proteínas forman el RISC (RNA-Induced
Silencing Complex, Complejo silenciador inducido por
ARN). RISC separa las piezas de ARN y las usa como guía para buscar y aparearse
(o "silenciar") toda secuencia de ARN que coincida con ellas. Al
bloquear este ARN, la célula se asegura que no se sinteticen proteínas
virales.
Si bien el siARN ha sido descubierto recientemente, el campo se ha expandido de
manera explosiva. Resulta claro que el siARN es un mecanismo molecular altamente
conservado en células eucariotas utilizado para controlar la expresión de los genes
durante el desarrollo embrionario y para defender sus genomas de invasores como
los virus a ARN y los transposones.
|
Silenciamiento Post-Transcripcional de un Gen, Iniciado por siARN. |
||||||||
|
|
||||||||
| El siRNA interacciona con la helicasa
(círculo púrpura) y la nucleasa (óvalo rosado) y forma un complejo
denominado RISC (RNA-induced silencing complex, Complejo
silenciador inducido por ARN) (1). La Helicasa del RISC usa ATP para
desdoblar el siARN, permitiendo que la cadena antisentido (cadena
corta en azul) se complemente con su "blanco" en el ARN mensajero (ARNm,
cadena larga en rojo) (2). La nucleasa del RISC corta el ARNm (3),
que finalmente es rápidamente degradado por otras ARNasas (4). En plantas y en Drosophila, DICER, un complejo que contiene una endonuclesa con actividad de helicasa y quinasa (óvalo verde) y una ARN dependiente, ARN polimerasa ( P roja en el triángulo verde), amplifica el siARN.
Modificado de: The New England Journal of Medicine,Volume 347:1364-1367, Volume 347:1364-1367, Number 17 |
|
|
Vea la Animación de una ribo-llave |
|
 |
 |
|
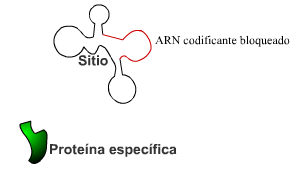
Fig. 2 |
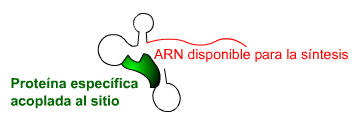
Fig. 3 |
Las ribo-llaves se pliegan dando orígen a figura compleja (Fig. 2). Una parte de la ribo-llave plegada puede recibir a una proteína específica que se amolda al sitio. La otra parte contiene el ARN que codifica a la proteína a sintetizar. La ribo-llave se "enciende" por acción de la proteína específica y permite que el ARN codificante sea traducido a proteína (Fig.3).
Los anticuerpos son proteínas globulares complejas fabricadas por
los organismos multicelulares como respuesta a la presencia de un antígeno
(una sustancia extraña al organismo). Las células que fabrican
los anticuerpos son los linfocitos, miembros de los glóbulos blancos
de la sangre. Los anticuerpos contribuyen a inmovilizar y destruir los
antígenos específicos. Un ratón puede fabricar cerca
de 10.000.000 de anticuerpos, es decir mas del total de sus genes. Las
proteínas de los anticuerpos están compuestos de dos cadenas largas
y dos cortas. Cada especie tiene una región constante característica
de la misma y del tipo de anticuerpo. La otra región es la variable,
en la que se encuentran los aminoácidos que dan la especificidad
al anticuerpo.
Susumu Tonegawa comprobó una vieja hipótesis que señalaba
que las regiones constantes y variables de los anticuerpos eran codificadas
por genes diferentes. Sus trabajos también mostraron que en los embriones
se reordenan fragmentos de los genes para dar origen a genes de la región
variable funcionales.
Los virus que afectan a los eucariotas son similares a los de los procariotas. Algunos de los virus a ADN pueden iniciar una infección (ídem al ciclo lítico de los procariotas) o formar provirus. Los provirus son ADN viral integrado al del huésped (ídem ciclo lisogénico). El virus de los monos (Simian Virus 40 o SV40) causa cáncer en los hámsters pero no en sus huéspedes normales. El SV40 puede, al igual que otros virus, introducir nuevos genes funcionales en su hospedador.
Los retrovirus pueden
también insertar su ácido nucleico en el ADN del huésped vía la transcriptasa reversa tal como fue mencionado. La transcriptasa reversa es introducida con el ARN dentro de
la célula huésped. En el proceso de fabricar la hebra de ADN complementaria
del ARN viral, la enzima también fabrica largas secuencias terminales
repetitivas ( del inglés: LTRs) al extremo terminal del ADN. Estas LTR hacen más fácil
la inserción en el ADN huésped. El ADN viral insertado fabrica ARN
viral y los necesarios para empaquetarlo en sus cubiertas de proteína
junto con la transcriptasa reversa.
El ADN viral, dependiendo de su inserción puede causar mutaciones
en el ADN del huésped, la mayor parte de ellos no lo hacen. Por lo contrario
se integran al genoma huésped y pueden pasar de generación en generación
si se las arreglaron para infectar las células de la línea
germinal. Del 0,5 al 1% del genoma del ratón podría ser de origen viral.
En realidad los elementos transponibles de los eucariotas, que recuerdan
a los de los procariotas en muchos sentidos, fueron descubiertos mucho antes
de que se los describiera en procariotas (década del 60) fruto de
los notables estudios de Barbara McClintock realizados en 1940 acerca de
los elementos transponibles del maíz, muchos años mas tarde (¿demasiados?)
se le
otorgó el Premio Nóbel.
Muchos transposones eucariotas primero son copiados a ARN y luego vuelta
a ADN en una localización diferente.
El cáncer es una enfermedad en las que las células escapan a los
controles normales de crecimiento. El cáncer es una enfermedad heredable
(al menos de célula madre a célula hija). Una vez que una
célula se vuelve cancerosa, todas las células que de ella
se originan son cancerosas.
A menudo son visible anomalías cromosómicas en las células
cancerosas. Muchas substancias carcinogénicas (actores que promueven
el cáncer) son también mutagénicas
(factores que producen mutaciones).
Los oncogenes aparentan ser genes normales pero algo anduvo mal en ellos
ya que promueven el cáncer.
Los virus parecen poder producir cáncer de tres formas. La presencia
del ADN viral podría alterar las funciones del ADN del huésped.
Las proteínas necesarias para la replicación del virus también afectarían la regulación de los genes del huésped. Dado que
la mayor parte de los virus que producen cáncer son retrovirus, los mismos
pueden ser vectores para la inserción de oncogenes.
La transferencia de genes entre células eucariotas permitirán a los médicos, que hasta ahora atacan la enfermedad a nivel fenotípico, hacerlo a nivel genotípico. El virus SV40 ha sido usado para inyectar el gen de beta globina en monos. Los virus sirven como posibles vectores para introducir alelos sanos en reemplazo de los enfermos.
|
ADN (ácido desoxirribonucleico) |
Un ácido nucleico compuesto de dos cadenas polinucleotídicas que se disponen alrededor de un eje central formando una doble hélice, capaz de autorreplicarse y codificar la síntesis de ARN.
Lugar donde esta "depositada" la información genética.
Ácido nucleico que funciona como soporte físico de la herencia en el 99% de las especies. La molécula, bicatenaria, esta formada por dos cadenas antiparalelas y complementarias entre sí. Su unidad básica, el nucleótido, consiste en una molécula del azúcar desoxirribosa, un grupo fosfato, y una de estas cuatro bases nitrogenadas: adenina, timina, citosina y guanina. Fórmula
|
ARN(ácido ribonucleico): Ácido nucleico formado por una cadena polinucleotídica. Su nucleótido, consiste en una molécula del azúcar ribosa, un grupo fosfato, y una de estas cuatro bases nitrogenadas: adenina, uracilo, citosina y guanina. | |
|
ARN polimerasa: Complejo enzimático que cataliza la síntesis de RNA (transcripción) utilizando como molde la cadena de una molécula de DNA. | |
|
Alelo ( del griego allelon = el uno al otro, recíprocamente): Formas alternativas de un gen, se hereda separadamente de cada padre (p.ej. en el locus para el color de ojos puede haber un alelo para ojos azules o uno para ojos negros. Uno o más de los estados alternativos de un gen. | |
|
Bacteriófago (del griego bakterion = pequeño bastón; phagein = comer): Literalmente "comebacterias". Virus cuyo huésped natural es una bacteria. Virus parásito de células bacterianas. | |
|
Bases apareadas (en inglés base pair, bp) Dos bases nitrogenadas (adenina y timina o guanina y citosina) mantenidas juntas por enlaces débiles (puente hidrógeno). Las dos hebras del ADN se mantienen juntas formando una doble hélice por los enlaces de sus bases apareadas. | |
|
Cromosomas(del griego chroma = color; soma = cuerpo): Estructuras del núcleo de la célula eucariota que consiste en moléculas de ADN (que contienen los genes) y proteínas (principalmente histonas). | |
|
Catalizador(del griego katalysis = disolución): Sustancia que disminuye la energía de activación de una reacción química, acelerando la velocidad de la reacción. | |
|
Cromatina: Material formado por ácidos nucleicos y proteínas que se observa en el núcleo de la célula en interfase | |
|
Crossing-over (del inglés entrecruzamiento): Proceso que ocurre en la meiosis e incluye la ruptura de un cromosoma materno y uno paterno (homólogos), el intercambio de las correspondientes secciones de ADN y su unión al otro cromosoma. Este proceso puede resultar en un intercambio de alelos entre cromosomas. | |
|
Enzima (del griego en = en; zyme = levadura): Molécula de proteína que actúa como catalizador en las reacciones bioquímicas. | |
|
Escherichia coli: Bacteria común, habitante del intestino humano, estudiada intensivamente por los genetistas en razón de su facilidad para crecer en el laboratorio, lo pequeño de su genoma y su falta de patogenicidad (generalmente..) | |
|
Eucariotas (del griego eu = bueno, verdadero; karyon = núcleo, nuez): organismos caracterizados por poseer células con un núcleo verdadero rodeado por membrana. El registro arqueológico muestra su presencia en rocas de aproximadamente 1.200 a 1.500 millones de años de antigüedad | |
|
Familia de Genes: Grupo de genes muy relacionados que fabrican productos similares. | |
|
Genes (del griego genos = nacimiento, raza; del latín genus = raza, origen): segmentos específicos de ADN que controlan las estructuras y funciones celulares; la unidad funcional de la herencia. Secuencia de bases de ADN que usualmente codifican para una secuencia polipeptídica de aminoácidos. | |
|
Haploide (del griego haploos = simple, ploion = nave): Célula que contiene solo un miembro de cada cromosoma homólogo (número haploide = n). En la fecundación, dos gametos haploides se fusionan para formar una sola célula con un número diploide ( por oposición, 2n) de cromosomas. | |
|
"Homebox" genes: Corta secuencia de nucleótidos virtualmente idéntica en todos los genes de los organismos que la contienen. Se la ha encontrado en numeroso organismos, desde la mosca de la fruta a los seres humanos. En la mosca de la fruta parecen determinar los grupos particulares de genes que se expresan durante el desarrollo. | |
|
Intrón: La secuencia de bases de ADN que interrumpe la secuencia de un gen que codifica para una proteína, esta secuencia se transcribe al mARN pero en un proceso de "corte y empalme" se separa del mismo antes que el mARN sea traducido a proteína. | |
|
Mutación (del latín mutare = cambiar): El cambio de un gen de una forma alélica a otra, cambio que resulta heredable. | |
|
Procariotas (del latín pro = antes, del griego karyon = núcleo, nuez): Tipo de célula que carece de núcleo rodeado por membrana, posee un solo cromosoma circular y ribosomas que sedimentan a 70 S (los de los eucariotas lo hacen a 80S). Carecen de organelas rodeadas por membranas. Se consideran las primeras formas de vida sobre la Tierra, existen evidencias que indican que ya existían hace unos 3.500.000.000 años | |
|
Retrovirus (del latín retro = girar hacia atrás): Virus que contienen una sola hebra de ARN como material genético, se reproducen copiando el ARN en ADN complementario usando la transcriptasa reversa. La hebra de ADN es luego copiada y, el ADN bicatenario, es insertado en el ADN de la célula huésped. | |
|
Transcriptasa reversa: Enzima utilizada para su replicación por los retrovirus; copia el ARN del retrovirus en una hebra complementaria de ADN. DNA-polimerasa RNA-dependiente, es decir, complejo enzimático que sintetiza una molécula de DNA tomando un RNA como molde. | |
|
Virus (del latín virus = veneno): Agente infeccioso de naturaleza obligatoriamente intracelular para sintetizar su material genético, ultramicroscópico y ultrafiltrable. Constan de un ácido nucleico (ADN o ARN) y un recubrimiento proteico. Entidad no celular, de muy pequeño tamaño. En estado extracelular son inertes. | |
|
Oncogén (del griego onkos = tumor): gen asociado al desarrollo del cáncer. Muchos oncogenes están directa o indirectamente relacionados con el control de la división celular. | |
|
Polimerasa (ADN o ARN): Enzimas que catalizan la síntesis de ácidos nucleicos en base a templados preexistentes, utilizando ribonucleótidos para el ARN y desoxirribonucleótidos para el ARN. | |
|
Promotor: Sitio del ADN donde se pegará la ARN polimerasa para iniciar la transcripción. | |
|
Producto génico: El material bioquímico, ya sea ARN o proteína, resultante de la expresión de los genes. La cantidad de producto génico se utiliza para medir cuan activo es un gen, cantidades anormales pueden correlacionarse con alelos que causan enfermedades. | |
|
Tecnología del ADN recombinante: Procedimientos utilizados para unir segmentos de ADN en un sistema libre de células ("cell free system" es decir fuera de un organismo o célula). Bajo condiciones adecuadas, una molécula de ADN recombinante puede introducirse dentro de una célula para ser replicado, ya sea autónomamente o integrada al cromosoma celular. |
![]()
Traducción, redacción y diagramación a cargo de :
Dr. Jorge S. Raisman, Dra. Ana María Gonzalez
Basada en la traducción de: gened.emc.maricopa.edu/bio/bioBioBookPROTSYN.html
Actualizado martes, 05 de noviembre de 2013 . Reproducción autorizada únicamente con fines educativos citando su origen. Se agradecen comentarios y sugerencias.
|