La estructura del genoma mitocondrial es circular como es el del genoma bacteriano. Se trata de una molécula circular de ADN, helicoidal, con doble hebra, y supercondensada. Se conocen también algunos pocos casos de genomas mitocondriales de forma lineal. En muchos casos, el contenido de GC (guanina-citosina) del ADNmt difiere en gran medida del ADN nuclear, y esto permite separar el ADNmt del nuclear por centrifugación en un l gradiente de cloruro de cesio. No existen histonas u otras proteínas semejantes asociadas al ADNmt. Existen muchas copias del genoma mitocondrial en cada mitocondria, las que se ubican en ciertas regiones llamadas nucleoides. En muchos animales, las dos hebras que componen el ADNmt difieren en densidad porque las bases nitrogenadas no están distribuidas de forma equilibrada en ambas hebras. Esto hace que una hebra sea más "pesada" y otra más "liviana". en el ADNmt. El contenido de genes de genomas mitocondriales de distintas especies es bastante similar tanto en número como en cantidad de funciones distintas. Sin embargo, el tamaño varía enormemente entre distintos organismos.
En los animales, el genoma mitocondrial generalmente es menor a 20 kb (kilobases); por ej. en el hombre el ADNmt tiene 16.569 bp (pares de bases). Por su parte, el ADNmt de una levadura contiene cerca de 80.000 bp (80 kb), mientras que en las plantas varía entre 100 y 2.000 kb. La mayor diferencia entre animales, hongos y plantas es que en los animales el ADNmt codifica algún producto en casi toda su extensión, mientras que en el genoma mitocondrial de hongos y plantas existen largas secuencias de DNA que no codifican productos. Los genes del ADNmt y su transcripción
|
 |
|
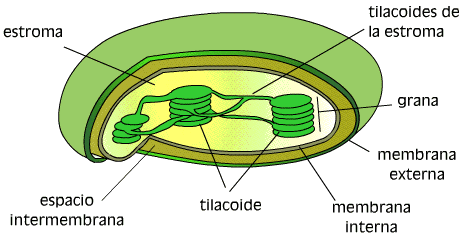
Esquema de un cloroplasto |
La estructura del genoma del cloroplasto es similar al mitocondrial. Aquí también el ADN tiene forma circular, está constituido por una doble hebra supercontraída y no existen proteinas como es el caso de las histonas de los cromosomas nucleares. Muchas veces existe una gran diferencia en el contenido de guanina-citosina del ADNcp en relación tanto al DNA nuclear como al ADNmt, lo que permite separar el ADNcp en un gradiente de cloruro de cesio. El ADNcp es una molécula más grande que el DNAmt de los animales, con un tamaño que varía entre 80 y 600 kb. Por ejemplo, el ADNcp del arroz contiene 155.844 pares de bases. Todos los genomas del cloroplasto que se conocen hasta ahora tienen una proporción muy alta de secuencias de ADN que no codifican ningún producto. El número de copias del ADNcp en cada cloroplasto es variable, pero siempre hay varias copias por cada cloroplasto y estas copias se distribuyen en grupos que forman nucleoides. La organización de los genes en el ADNcp. El genoma cloroplástico contiene los genes para producir cada uno de los ARNr de los ribosomas típicos del cloroplasto (16S, 23S, 4,5S y 5S). También contiene genes para los ARNt, y genes que codifican algunas (pero no todas) las proteínas requeridas en los procesos de transcripción y traducción dentro del cloroplasto (como ser las proteínas de los ribosomas, las subunidades de la ARN polimerasa y los factores de traducción), o requeridas para la fotosíntesis. Algunos, aunque no todos los genes que codifican proteínas en el ADNcp transcriben intrones. Algunas de las proteínas con funciones dentro del cloroplasto son codificadas en el DNA nuclear y sintetizadas en el citoplasma y luego ingresadas al cloroplasto. En la Figura 2 se esquematiza la organización de los genes en el ADNcp.
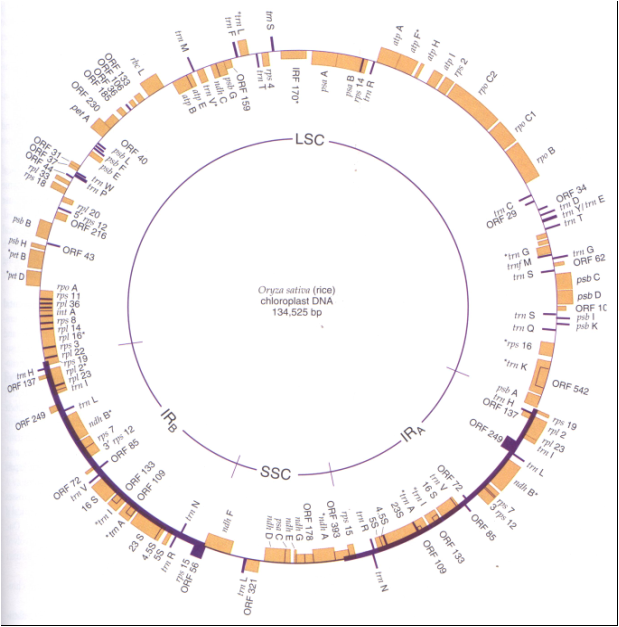
Figura
2: Organización del genoma del cloroplasto del arroz
(Oryza sativa)
De manera característica, el genoma del cloroplasto contiene dos copias de cada uno de los genes para la producción de ARN de transferencia (ARNt). Los dos sets de genes de ARNt se localizan en dos regiones de 10 a 25 kb con secuencias repetitivas idénticas pero con orientación invertida que se conocen como IRA e IRB (Fig. 2). Hay otros genes en estas secuencias repetitivas invertidas y por lo tanto esos genes también están repetidos. La ubicación de estas secuencias repetitivas definen otras dos regiones del genoma donde los genes no están repetidos: una región corta SSC (short single copy) y una región más larga llamada LSC (long single copy). Tanto en tabaco como en arroz, dos especies para las cuales se conoce el ADNcp, existen 30 genes de ARNt, mientras que en Marchantia (una hepática) son 32. Se han identificado cerca de 100 secuencias ORF (open reading frames) que se supone que codifican proteínas. Aproximadamente 60 de esas ORFs ya han sido correlacionadas con genes que codifican proteínas con función conocida, mientras que el resto no se sabe aun que función cumplen. La síntesis de proteínas dentro del cloroplasto se realiza en ribosomas específicos del cloroplasto que son de 70S con dos subunidades de 50S y 30S. Las subunidad de 50S contiene una copia de cada una de las moléculas de ARNr de 23S, 5S y 4,5S y la subunidad 30S contiene una molécula de ARNr de 16S. No se sabe muy bien aun cuántas proteínas constituyen cada una de las subunidades ribosómicas, pero sí se sabe que algunas de esas proteínas son codificadas por ADN nuclear y otras por ADNcp.
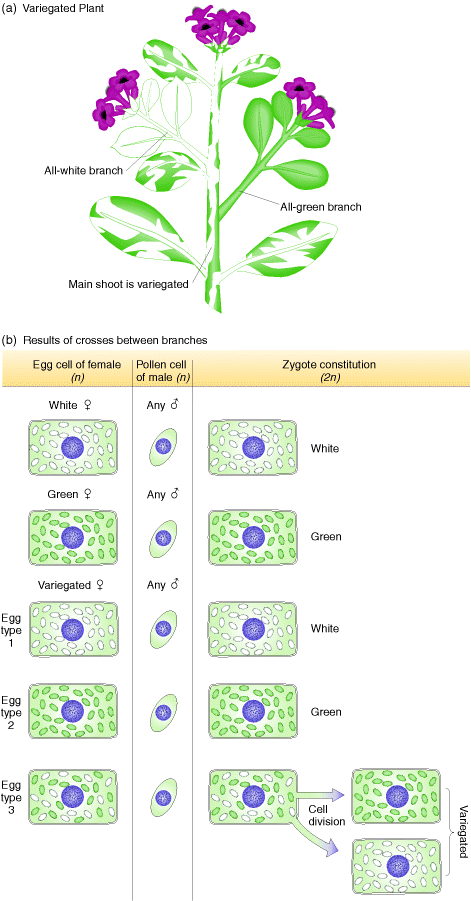
Existen características fenotípicas que se transmiten exclusivamente vía materna y están controladas por genes citoplasmáticos. Un ejemplo ya clásico es el de las hojas variegadas (verdes con manchas blanco-amarillentas) de planta ornamental Mirabilis jalapa, En 1909, Carl Correns publicó algunos resultados sorprendentes de una serie de hibridaciones que practicó en esta especie. Existen algunas plantas variegadas en las que la mayoría de las ramas son variegadas, aunque también existen ramas totalmente verdes y otras totalmente blanco-amarillentas (de ahora en mas diremos solamente "blancas" para simplificar). Las flores se forman sobre los tres tipos de ramas, y Correns hizo todos los cruzamientos posibles entre las distintas clases de ramas, castrando las flores que actuarían como madres y tomando polen de aquellas ramas que actuarían como progenitores masculinos. Los resultados son los del siguiente cuadro:
| Fenotipo de la rama que actuó como madre | Fenotipo de la rama que aportó el polen | Fenotipo de la progenie |
| blanco | blanco | blanco |
| blanco | verde | blanco |
| blanco | variegado | blanco |
| verde | blanco | verde |
| verde | verde | verde |
| verde | variegado | verde |
| variegado | blanco | variegado verde o blanco |
| variegado | verde | variegado, verde o blanco |
| variegado | variegado | variegado, verde o blanco |
Imagen
tomada de Griffits
En primer lugar, hay que destacar en estos resultados la diferencia entre cruzamientos recíprocos: por ejemplo, blanco X verde no da el mismo resultado que verde X blanco. En general, el fenotipo de la madre es el único responsable del fenotipo de la progenie. El fenotipo del polinizador es irrelevante respecto al fenotipo que se obtiene en la progenie. El único que contribuye al fenotipo de la progenie es el progenitor femenino. Es un caso característico de herencia materna.
Ciertamente, la progenie blanca no puede sobrevivir porque carece de clorofila, pero las demás progenies sobreviven y pueden ser utilizadas para hacer nuevos cruzamientos. Siempre las progenies siguen el mismo patrón de herencia materna observada en el experimento original.
Los resultados de Correns se deben al hecho de que en la fecundación, el gameto masculino no aporta cloroplastos porque es el gameto femenino, la ovocélula, la que aporte prácticamente todo el citoplasma para formar el cigoto.
Los plástidos (cloroplastos y/o leucoplastos) para el nuevo individuo son aportados por la ovocélula. En los dos primeros tipos de cruzamientos donde el progenitor femenino era blanco o verde, la herencia era estrictamente materna: cuando la madre es blanca, la ovocélula aporta solamente leucoplastos; cuando la madre es verde, la ovocélula lleva solo cloroplastos. Sin embargo, cuando la madre es variegada, la ovocélula puede llevar solamente cloroplastos y entonces la nueva planta será verde; si la ovocélula lleva solo leucoplastos, la progenie será blanca (y morirá por falta de clorofila); pero si la ovocélula lleva una mezcla de leucoplastos y cloroplastos, la progenie será variegada.
Hay que tener en cuenta, sin embargo, que en muchos casos donde en una célula existe una mezcla de plástidos genéticamente diferentes (leucoplastos y cloroplastos) por lo común suele observarse que al dividirse la célula por mitosis, funciona un proceso de agrupamiento mediante el cual una de las células hijas lleva los plástidos verdes y otra los blancos. En la mayoría de las plantas los orgánulos son heredados a través del progenitor femenino aunque existen algunos casos, raros por cierto, donde la herencia de los orgánulos es a través del progenitor masculino.
En plantas superiores la esterilidad masculina se debe a una falla en la formación de polen, a la producción de polen no funcional, o a la incapacidad de ciertas plantas para producir la dehiscencia de las anteras en la floración.
La androesterilidad puede estar determinada genéticamente por factores nucleares, generalmente de características recesivas. Sin embargo, la androesterilidad puede deberse también a genes extranucleares.
Existe un tipo de androesterilidad controlada genéticamente por factores citoplásmicos y nucleares que interactúan. En este caso existe un factor citoplásmico que produce androesterilidad y se transmite por la ovocélula. O sea, de acuerdo al citoplasma, una planta puede ser normal (genotipo citoplásmico N) o androestéril (genotipo citoplásmico S), Sin embargo en el núcleo pueden existir factores que eventualmente restauren la fertilidad. El genotipo nuclear, como vimos hasta ahora para cualquier gen nuclear, puede ser homocigota dominante para fertilidad (Rf Rf homocigota para esterilidad (rf rf) o heterocigota (Rf rf). Relacionando genes citoplásmicos con genes nucleares podemos tener las siguientes combinaciones genotípicas:
|
N / Rf Rf N / Rf rf N / rf rf |
S / Rf Rf S / Rf rf S / rf rf |
Siempre que el citoplasma sea normal, la planta va a presentar un genotipo androfértil. Por el contrario, si el citoplasma tiene el factor de androesterilidad S, la planta será macho-estéril, a no ser que en el núcleo exista el alelo dominante (Rf) para restaurar la fertilidad. Por lo tanto, solamente las plantas con genotipo S /rf rf son androestériles.
Por la herencia materna del factor citoplásmico, las semillas de una planta con citoplasma N siempre dará descendientes androfértiles, mientras que la semilla cosechada de una planta con citoplasma S podrá producir tres tipos de progenies, dependiendo de su propio genotipo nuclear y del genotipo nuclear del polinizador:
1) toda la progenie androfértil, Una madre S Rf Rf dará siempre descendientes androfértiles independientemente del polinizador que usemos.
2) toda
la progenie androestéril: una madre androestéril S rf
rf con un polinizador N rf rf tendrá el 100% de la
progenie androestéril (S rf rf
Por eso, a una línea
con genotipo N rf rf se la llama
línea mantenedora ( o
línea M) porque usada como fuente de polen sobre
plantas androestériles se mantendrá en la descendencia
el genotipo androestéril S rf rf
3) una mezcla de plantas androfértiles y androestériles. Aquí dependerá tanto del genotipo materno como del paterno.
Si escribimos en primer término al progenitor femenino y a la derecha el masculino, los cruzamientos posibles de una planta con citoplasma S (además de los expuestos en 1 y 2, sería:
S Rf rf X N Rf Rf ó S Rf Rf
...........progenie 100% androfértil
S Rf
rf X N Rf rf
ó S Rf rf...............progenie 75% androfértil y 25%
androestéril
S Rf rf X N rf rf...............................progenie
50% androfértil y 50% androestéril
S rf rf X
N Rf Rf
ó S Rf Rf..............progenie 100% androfértil
S
rf
rf X N Rf rf ó S Rf rf..................progenie 50%
androfétil y 50% androestéril
Por esta razón se llama línea restauradora de la fertilidad ( o línea R) a aquella que sea homocigota dominante en su constitución nuclear (N Rf Rf ó S Rf Rf), ya que siempre que la usemos como fuente de polen sobre una planta androestéril, la fertilidad se restaurará en el 100% de la progenie.
Este sistema de androesterilidad controlada por interacciones entre factores nucleares y citoplásmicos es la principal herramienta en la producción de híbridos comerciales y en su momento revolucionó el mejoramiento genético especialmente del maíz.
Molecular
Biology of the Cell. 4th ed. Alberts,
Bruce; Johnson, Alexander; Lewis, Julian; Raff,
Martin; Roberts, Keith; Walter, Peter
New York: Garland
Publishing; 2002.
Modern
Genetic Analysis. Griffiths, Anthony J.F.;
Gelbart, William M.; Miller, Jeffrey H.; Lewontin,
Richard C.
New York: W.
H. Freeman & Co.; c1999
Nota: Tradución parcial y adaptación de iGENETICS de Peter J Russell. Pearson Education Inc., publishing as Benjamín Cummings. 2001, realizada por el Prof. Camilo Quarin, Cátedra de Genética de la Facultad de Ciencias Agrarias. UNNE (2003)
|
|