Genética humana

El genoma humano | Mas allá del Proyecto Genoma Humano
| El
cariotipo humano | Cromosomas X e Y| Anormalidades
cromosómicas humanas | Desórdenes
alélicos en humanos (recesivos) | Desordenes alélicos
en humanos (recesivos) en "genes solo a ARN" | Desórdenes
alélicos en humanos (dominantes) | Caracteres
ligados al sexo | Diagnostico
de las enfermedades genéticas humanas | Sondas
radioactivas | Misceláneas
| Glosario
| Enlaces | Índice

[ Principal ] [ Arriba ] [ Introducción a la genética ] [ Interacción genética ] [ Genética humana ] [ X vs. Y ] [ Genoma Extranuclear ] [ Mutación ] [ Glosario de Genética ]

En febrero del 2001 Nature
publicó la versión del genoma humano dada a
conocer por el HGP
(Proyecto genoma humano) cuyo director es Francis Collins y
Science,
la versión producida por el grupo de
Craig Venter (®
 )
ambas con las correspondientes versiones digitales. )
ambas con las correspondientes versiones digitales.
La secuencia asciende a unas 2.100 millones de bp y deriva del
estudio de la eucromatina.
La
heterocromatina (que representa aproximadamente un 5% del total) y que es difícil de
secuenciar, posiblemente contenga algunos
genes. En ambas publicaciones se detallan los métodos utilizados, las
perspectivas y significado de la secuenciación para la ciencia y la humanidad.
Recordemos que en eucariotas las regiones codificantes de un gen se encuentran
fragmentadas en pequeñas
piezas denominadas exones,
separadas por largas secuencias de ADN no codificantes. Solo cuando el ARNm
es editado, durante el proceso
de transcripción, los exones se juntan.
Collins señala que para identificar los genes
funcionales los científicos dependen de una variedad de indicios.
Algunos surgen de la comparación con las bases de datos de
ADNc
(complementario), que son
copias exactas de los ARNm. También ayuda la comparación con el ya
secuenciado genoma de
la rata en razón de que los genes de la rata y de los humanos son a menudo
muy parecidos. Sus secuencias se encuentran conservadas en ambos
genomas, mientras que no sucede lo mismo
en una gran cantidad de ADN que los rodea.
Cuando estos (u otros) indicios no están disponibles los científicos
dependen para la identificación exclusivamente de las predicciones realizadas
en computadoras ("gene-predicting computer
algorithms").
- Los resultados señalan la existencia de entre 30.000 y 40.000
genes,
solo aproximadamente el doble de los de Drosophila melanogaster.
Esta cifra esta en el orden de lo pronosticado en base a la secuenciación
del cromosoma 21 y resulta
ser notablemente inferior a los
140.000 estimados en base a la complejidad
funcional del ser humano.
- Los voceros del proyecto aluden a las
diferentes formas en la que puede introducirse complejidad en
un sistema por mecanismos se dan en humanos y en
otros genomas de organismos superiores a saber:
- Proteínas multifuncionales
- Cortes y ensambles alternativos durante el proceso de edición
del ARNm (al menos un tercio de los genes humanos producen diferentes proteínas
por medio de ediciones alternativas del mensaje ("alternative splicing"
) codificado en el "pre ARNm".
- Como los algoritmos nos son un 100%
fiables (a veces "ven" genes donde no hay tales, o pasan por alto
algunos de los existentes) algunos científicos dudan del contaje
realizado, entre ellos, William Haseltine ( el CEO de
Human Genome
Sciences considera que existen mas del doble de genes de los
reportados por los dos grupos
- Causa cierta perplejidad encajar el significado de la presencia de unos
200 genes
bacterianos que "invadieron" el "futuro genoma humano"
en alguna etapa evolutiva muy primitiva (millones de años atrás) producto
de una transferencia horizontal de
genes.
- El genoma contiene grandes regiones pobres en genes ("desiertos").
Mas de la mitad del genoma humano consiste en secuencias repetitivas
sin función conocida ("junk DNA" o "ADN basura") destacándose que el cromosoma 19
que tiene un 57% de ADN repetitivo.
La mayor parte del mismo deriva de la actividad parasitaria
de transposones que se replicaron e insertaron copias de si mismos en otros
sitios. Actualmente las diferentes familias de transposones
parecen haber dejado de "vagar" por el genoma y solo quedan
en el sus "fósiles". Unos 50 genes parecen haber sido originados
en transposones lo cual sugiere un cierto rol protagónico en la evolución
del genoma.
- Sumándose a la repetición de secuencias causada por los
transposones grandes segmentos, tanto dentro como entre cromosomas del genoma,
parecen haberse duplicado a lo largo de los tiempos. Los investigadores
creen que esto permitió a la evolución "realizar ensayos" con
los mismos sin destruir la función original y probablemente permitió la
expansión de muchas familias de genes humanos.
- Tanto el HGP como Celera identificaron en el ADN una multitud
de posiciones de bases que difieren entre individuos los llamados
"snips" o SNPs (single polynucleotide polymorphism).
HGP si bien el 99,9% de las secuencias son idénticas en todos los
individuos el aproximadamente 0,1%, que corresponde a los SNPs,
difiere de uno a otro. Se espera aprender de
ellos como los genes hacen diferentes a los individuos y, en
particular, la razón por la cual algunos son mas susceptibles a ciertas
enfermedades.
Celera utilizó par la secuenciación el ADN de cinco individuos (un latino, un
asiático, dos caucásicos y un afroamericano) y las cinco
secuenciaciones fueros coincidentes en un 99.9%.
Una rumor se ha esparcido: uno de los caucásicos seria
el propio Craig Venter.
Los avances realizados en este campo fueron espectaculares y, mas
allá de los informes que parecen destinados a impactar a la opinión
pública (en especial inversores actuales y futuros..), resta
todavía una enorme cantidad de trabajo a realizar: completar la ya
señalada secuenciación de la heterocromatina, rellenar los "huecos"
("gaps") dejados en secuencias muy difíciles, encontrar la
totalidad de los genes que codifican para proteínas y aquellos que solo
codifican para ARN, descubrir secuencias regulatorias, "blancos"
donde las moléculas interactúan con lo genes e identificar mas funciones
de los mismos, sin dejar de lado las necesarias revisiones para la
corrección de los errores que pudieron escaparse en estas primeras
versiones.
2004, Mas allá del Proyecto Genoma Humano
| Sydney
Brenner del Instituto
Salk, Nobel 2002 escribió, "Los antiguos genetistas
conocían de lo que estaban hablando cuando utilizaban el
término "gene", pero el término parece haber sido
"corrompido" por la moderna genómica pasando a
significar cualquier segmento de una secuencia que se
expresa. |
| Claes
Wahlestedt del Instituto
Karolinska (Suecia, Sweden,) nos dice "tendemos
a no hablar mas acerca de "genes“ ; nos referimos a
cualquier segmento que es trascripto [a ARN] como una
"unidad trascripcional" . |
|
Carmen
Sapienza de la Universidad
de Temple (casi emitiendo un nuevo postulado de la ley
de Murphi aplicable al genoma) nos dice: "todo aquello que
pueda ocurrir en el genoma, sucederá"
|
El genoma, la suma de la información heredable contenida
en los cromosomas y que gobierna el desarrollo de un organismo, no es
un texto estático pasado de una generación a otra, en vez de
ello el genoma es una maquinaria bioquímica de extraordinaria
complejidad que, como cualquier maquinaria, opera en el espacio
tridimensional y esta constituido por partes diferentes e
interactuantes. Estas partes conformas por lo menos tres niveles de
información.
El 98% del genoma que ha sido considerado durante mucho tiempo
como "silencioso", en el mejor de los casos , o como
"basura" (junk) en muchos otros, no se estudió en profundidad en general por falta de
métodos
adecuados. En estos últimos años los genetistas han
explorado las partes no visibles del genoma en busca de
explicaciones a anomalías que que contradicen el dogma central de
la biología: enfermedades familiares que se manifiestan de manera
impredecible (aún en gemelos.....) o el "encendido y apagado" de genes en canceres
que no presentan mutaciones. Estos estudios se centraron en los otros dos niveles
adicionales de información encontrándose que se encuentran
profundamente relacionados con la herencia conectándola con el
desarrollo y las enfermedades. Existen nuevos esfuerzos colaborativos
que están tratando de examinar estas regiones tales como el
Proyecto ENCODE
( (Encyclopedia of
DNA Elements) y empresas particulares entre ella la pionera
Affymetrix que explora la organización y función de la
totalidad el Genoma Humano.
- La
primera de estas partes, los genes que codifican para proteínas (aproximadamente 30.000
regiones del Genoma Humano), conforman y representan alrededor de un 2%
del ADN total.
-
La segunda de estas partes, esta conformada (entre
otros) por los genes "solo a ARN" (RNA only genes)
a partir de los cuales no se sintetizan proteínas.
Estos genes
no convencionales dan origen a ARNs activos y por medio de
ellos alteran el comportamiento de "genes
normales".
La
tercera parte esta conformada por la información
epigenética guardada en las proteínas y otras
moléculas (por ej. grupos metilo) que se encuentran rodeando o esta pegadas al ADN.
Estas marcas epigenéticas pueden influir en las
características o en la salud de un individuo (algunas pasan de
padres a hijos) sin alterar
la secuencia del ADN.

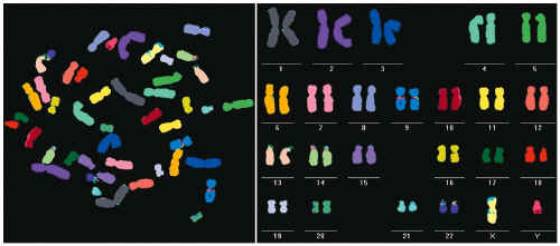
El cariotipo humano
Existen 44 autosomas
y 2 cromosomas sexuales (ver La
secuenciación del Cromosoma Y) en el genoma
humano lo que hace un total de 46 cromosomas.
Los cariotipos son fotografías
o representaciones gráficas de los cromosomas de una célula somática de un
organismo.
A grandes rasgos los pasos de la técnica para obtenerlos son los
siguientes:
-
toma de sangre periférica y
separación de los glóbulos
blancos
-
incubación en presencia de productos
que inducen a la mitosis (mitogénicos) tales como la fitohemoaglutinina
-
detención de la mitosis en la
metafase (utilizando colchicina,
que interfiere con los microtúbulos del huso)
-
un paso por un medio hipotónico
que hace que las células se
hinchen
-
depositar una gota de la preparación
entre porta y cubre (sobre el cual se hace presión)
-
fijar, colorear y fotografiar los
núcleos estallados
-
finalmente la foto se amplifica y
los cromosomas se recortan y se ordenan
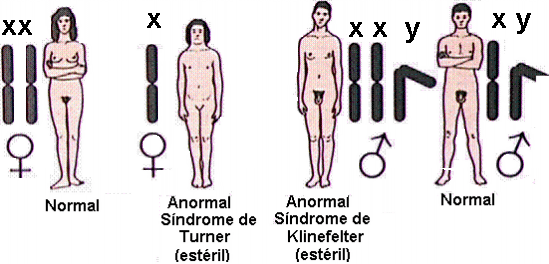
Anormalidades
cromosómicas humanas

-
Una anormalidad común es la causada
por la no disyunción ( la falta de segregación) de un cromosoma durante la
anafase II de la meiosis.
Un gameto al cual le falta un cromosoma no puede
producir un embrión viable pero ocasionalmente un gameto con n+1
cromosomas puede producir un embrión viable. En humanos la no disyunción
esta generalmente asociada al cromosoma 21, que produce una enfermedad
genética conocida como síndrome
de Down (o trisomía 21). Algunos casos son el resultado de una translocacíon
en el cromosoma de uno de los padres. La incidencia del Síndrome de Down
se incrementa con la edad de la madre, sin embargo un 25% de los casos son
el resultado de un cromosoma extra de origen paterno.
Entre los recientes avances en los estudios cromosómicos se
encuentra la secuenciación del cromosoma humano 21 (NATURE | VOL 405 |18
MAY 2000 | www.nature.com). El
cromosoma 21 es el más
pequeño de los autosomas. Una copia extra de este es la causa genética más
frecuente de retardo mental (el Síndrome de Down
con una incidencia de 1 cada 700 nacimientos). En el trabajo los autores describen la secuenciación y
catalogación del brazo largo del cromosoma 21. Secuenciaron 33.546.361
pares de bases (bp) del ADN con gran precisión (99.7% de cobertura restando
unas 100 Kb). También secuenciaron 281.116 bp del brazo corto: entre
las identificaciones realizadas se incluyen duplicaciones probablemente
relacionadas con anormalidades cromosómicas y estructuras repetitivas en las regiones teloméricas.
Encontraron por otra parte que el numero total de genes humanos parece estar
sobreestimado.
El cromosoma 21 representa el 1% del Genoma Humano o sea que si
el total fuera 100.000 era de esperarse que contuviera alrededor de 1.000
genes. Aunque muchos auguraban una pobreza de genes no se esperaba tanta pobreza
como la encontrada. El análisis del cromosoma 21 revela 127 genes
conocidos, 98 predecidos y 59
pseudogenes.
Los autores señalaron que si se considera que el cromosoma 22 tiene alrededor
de quinientos genes, entre el 21 y el 22 suman alrededor de 750 y, como ambos
constituyen el 2% del genoma humano, la extrapolación lleva a un Genoma
Humano de aproximadamente 40.000 genes Y NO DE 100.000.
Los detalles de los protocolos usados por los cinco centros
intervinientes que incluyen los métodos de construcción
de los mapas se encuentran en la WWW (mucho del software utilizado es de dominio publico o desarrollados
por el consorcio de instituciones intervinientes para permitir un análisis eficiente). La información se
encuentra en las siguientes paginas
-
Anormalidades en los cromosomas
sexuales pueden ser causadas por la no disyunción de uno o más
cromosomas sexuales. Cualquier combinación (tipo XXXXY) produce machos con
deficiencias. Hombres con mas de un cromosoma X son generalmente
deficientes mentales y estériles. Se conocen mujeres XXX y XO, siendo en
su mayor parte estériles.
-
Las deleciones en cromosomas se
asocian también a otros síndromes como el tumor de
Wilm.
 |
|
Modificado de: http://www.whfreeman.com/life/update/. |
La detección prenatal de las anormalidades cromosómicas se realizan por un
procedimiento llamado amniocentésis.
Desordenes alélicos
en humanos (recesivos)

- El
primer carácter Mendeliano en humanos, la braquidactilia
se describió en 1905. Actualmente se conocen mas de 3.500.
- El albinismo
es un caso de herencia Mendeliana . Los individuos homocigotos
recesivos
(aa) no fabrican melanina,
y por lo tanto tiene su cara, cabello, y ojos que son blancos o amarillos.
Los padres heterocigotos
con pigmentación normal (Aa), producen dos tipos de gametos: A o a. De la
cruza de dos heterocigotos 1 de 4 hijos puede resultar albino.
- La fenilcetonuria (PKU) es un
desorden hereditario recesivo en los que el individuo sufre por su
incapacidad para transformar el aminoácido fenilalanina en tirosina. Los
individuos homocigotas recesivos para este
 tienen un exceso de
fenilalanina y productos de su metabolismo en sangre y en la orina. Estos
metabolitos pueden ser dañinos para el sistema nervioso que se está
desarrollando y producen retardo mental. Uno de cada 15.000 recién nacidos
sufre este problema. La detección de esta enfermedad en el recién nacido
es hoy parte de la rutina bioquímica. Si usted examina con detalle un
producto que contenga Nutra-Sweet (un edulcorante artificial que,
bioquímicamente, es un péptido) verá que existe un aviso para aquellos que
sufren de PKU dado que la fenilalanina es unos de los aminoácidos que
forma parte de Nutra-Sweet. Los enfermos de PKU se someten a dietas
restringidas en fenilalanina, suficiente para las necesidades metabólicas
pero no para producir intermediarios dañinos.
tienen un exceso de
fenilalanina y productos de su metabolismo en sangre y en la orina. Estos
metabolitos pueden ser dañinos para el sistema nervioso que se está
desarrollando y producen retardo mental. Uno de cada 15.000 recién nacidos
sufre este problema. La detección de esta enfermedad en el recién nacido
es hoy parte de la rutina bioquímica. Si usted examina con detalle un
producto que contenga Nutra-Sweet (un edulcorante artificial que,
bioquímicamente, es un péptido) verá que existe un aviso para aquellos que
sufren de PKU dado que la fenilalanina es unos de los aminoácidos que
forma parte de Nutra-Sweet. Los enfermos de PKU se someten a dietas
restringidas en fenilalanina, suficiente para las necesidades metabólicas
pero no para producir intermediarios dañinos.
- La enfermedad de Tay-Sachs es autosómica
y recesiva, su evolución lleva a la degeneración del sistema nervioso. Los
síntomas se manifiestan luego del nacimiento. Los niños homocigotas para
el alelo recesivo raramente superan la edad de cinco años. Los enfermos no
tienen capacidad para fabricar una enzima (la N-acetil-hexosaminidasa),
que elimina un lípido (conocido como gangliósido GM2). Este lípido se
acumula en los lisosomas de las células cerebrales y, eventualmente las
mata. Si bien es rara en la población general (1 de 300.000 nacimientos),
fue (hasta hace poco) alta (1 en 3600 nacimientos) entre los descendientes
de judíos de Europa central. Se piensa que uno de cada 28 entre los judíos
americanos es un portador dado que el 90% migró de esa área. La mayor
parte de los bebés que nacen actualmente con Tay-Sachs se originan de
padres no judíos que no entran en los programas de control genético por no
estar prevenidos debido a su baja incidencia en poblaciones no judías.
- La hemocromatosis hereditaria es
autosómica y recesiva, se caracteriza por un exceso de depósitos de hierro
principalmente en el hígado y páncreas (hasta 100 veces la concentración
normal) que conducen a cirrosis, diabetes, artritis..., responde a una
terapia clásica: la sangría, por esta razón la mayoría de las mujeres
están a salvo hasta la menopausia dado que la menstruación elimina el
exceso de hierro. Se suele dar entre los Amish.
- La anemia drepanocítica, autosómica
y recesiva, es un ejemplo al que se recurre varias veces en este curso. El
9% de los negros norteamericanos son heterocigotos para esta mutación, y
un 0,2% homocigotos para ella. El alelo recesivo causa la sustitución de
un solo aminoácido en la cadena beta de la hemoglobina. Debido a la
mutación, cuando la concentración de oxígeno es baja la hemoglobina se
vuelve insoluble por lo cual los glóbulos rojos se deforman (falciformes
o en forma de hoz) tornándose muy frágiles (también ocurre el parásito que
produce la malaria, no puede desarrollarse en ellos. Aparentemente esta
mutación se originó en África, y resulta un interesante ejemplo de como la
selección natural puede mantener caracteres a primera vista desfavorables.
Los heterocigotos fabrican suficiente cadenas beta normales de hemoglobina
como para no sufrir los síntomas, mientras la concentración de oxígeno se
mantenga alta como ocurre a nivel del mar. La drepanocitosis ofrece un
buen ejemplo de las dificultades del empleo en ciertos casos de los
términos mendelianos de dominante y recesivo, pues en este caso si
hablamos del fenotipo de la drepanocitosis como una enfermedad, estamos
ante una enfermedad autosómica recesiva ya que es necesario que el
individuo sea homocigoto para tener la enfermedad (el heterocigoto es
asintomático). Sin embargo si se examina desde el punto de vista de la
hemoglobina que posee (analizada cromatográficamente) la clasificación
correcta sería decir que la herencia de la hemoglobina S es codominante
autosómica (como los grupos sanguíneos) ya que ambas hemoglobinas se
detectan en los
heterocigotos.
- La fibrosis quística o mucovicidosis
del páncreas es la enfermedad hereditaria autosómica y recesiva mas
frecuente en las poblaciones de raza blanca. Se caracteriza por la producción de un mucus espeso,
insuficiencia pancreática exógena y electrolitos altos en el sudor. En
ausencia de tratamiento es mortal y con tratamiento rara vez superan la
adolescencia. La causa es un defecto en un único gen que se encuentra en
el brazo largo del cromosoma 7. La proteína alterada es una proteína de
transporte de membrana que forma un canal iónico para el Cl. La causa mas
frecuente es una deleción de tres bases:
 , que provocan la falta de
fenilalanina en la posición 508 de la proteína de transporte, lo cual le
impide fijar ATP.
Una vez más necesitamos recurrir a hipótesis evolutivas para explicar la
alta incidencia de un alelo tan terrible en la naturaleza, pareciera ser
en este caso, que los heterocigotas son resistentes a la tifoidea,
enfermedad que en otros tiempos tenía una alta tasa de mortalidad, lo cual
explicaría esta "anómala selección". , que provocan la falta de
fenilalanina en la posición 508 de la proteína de transporte, lo cual le
impide fijar ATP.
Una vez más necesitamos recurrir a hipótesis evolutivas para explicar la
alta incidencia de un alelo tan terrible en la naturaleza, pareciera ser
en este caso, que los heterocigotas son resistentes a la tifoidea,
enfermedad que en otros tiempos tenía una alta tasa de mortalidad, lo cual
explicaría esta "anómala selección".
Desordenes alélicos
en humanos (recesivos) en "genes
solo a ARN"
- Hipoplasia del cartílago piloso (CHH,
cartilage hair hypoplasia), identificada por primera vez entre la
población Amish. Uno de cada 19 lleva una copia defectuosa del gen
recesivo, que causa no solo enanismo, sino además alta propensión a
cánceres y desórdenes inmunológicos
La razón de este "apartado" dentro de las enfermedades
genéticas recesivas es que una vez mas los Amish, fuente
aparentemente inagotable de sorpresas en genética, permitieron
al Genetista Maaret Ridanpää
de la Universidad de Helsinki
descubrir que el responsable de esta enfermedad es un gen
recesivo pero codificante solo para ARN, llamado RMRP. Un
cambio en solamente una base de este gen (la mutación mas usual es la
substitución de un G por A) resulta responsable de esta
enfermedad (cuando la mutación se da en ambos cromosomas) .
El ARN transcripto por este gen es capaz de interactuar con
proteínas ( no servir de mensaje para sintetizar una proteína!!!!) y
formar una enzima que altera la función de la mitocondria.
Desórdenes alélicos
en humanos (dominantes) Los
casos son raros, a pesar de que por definición se expresan siempre, la razón está
en la dificultad para reproducirse de los gravemente afectados.
-
| El enanismo acondroplástico
es uno de los trastornos autosómicos dominante más comunes. Se
caracteriza por las extremidades cortas y caracteres faciales
particulares.
A pesar que esta condición se debe a un autosomico
dominante, el 80% de los casos se deben a nuevas mutaciones
esporádicas. La mutación involucra el gene que codifica el
factor de crecimiento de los fibroblastos 3 (FGFR3), ubicado
en el cromosoma 4; es una mutación puntual que cambia el
aminoácido arginina por glicina |
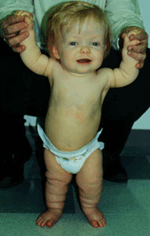
Imagen de
una niña.
Medical Genetics. 1999 |
- La enfermedad de Huntington es
autosómica y dominante, su resultado es la destrucción progresiva de la
células del cerebro. Si uno de los padres es heterocigoto (lo cual por
supuesto implica que es enfermo) el 50% de los hijos tendrá el carácter y
la enfermedad. Si es homocigota el 100%. La enfermedad no se manifiesta
hasta la edad de 30 años, aunque algunos fenómenos relacionados con la
enfermedad pueden presentarse a los 20 años.
La enfermedad, perteneciente el grupo de las enfermedades
neurodegenerativas producidas por expansión de trinucleótidos
- Polidactilia es la presencia de seis
dedos en manos o pies. Actualmente a menudo el dedo extra se extirpa quirúrgicamente
al nacer y el individuo no conoce que es portador del carácter. Una de la mujeres de Enrique VIII tenía un dedo extra . En
ciertas familias del sur de USA es mas común. El dígito extra raramente es
funcional y sin ninguna duda causa problemas en el momento de comprar
guantes.......
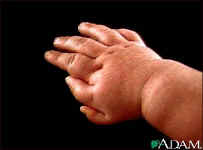
Imagen
de la mano de un niño con polidactilia. http://health.allrefer.com/
Caracteres ligados al sexo
- El daltonismo o ceguera a los
colores aflige al 8% de los hombres y al 0,04 % de las mujeres. La
percepción del color depende de tres genes, cada uno de los cuales produce
compuestos sensibles a diferentes partes del espectro visible. Los genes
para la detección del rojo y el verde se encuentran en el cromosoma X. La
detección del azul es autosómica.
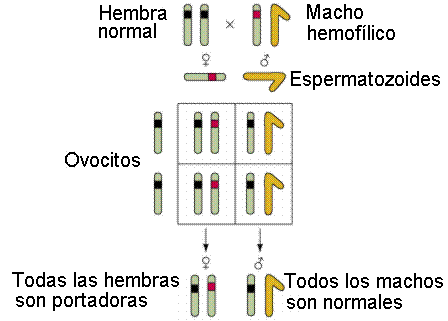
- Por hemofilia
se conoce a un grupo de enfermedades en las cuales la sangre no coagula
normalmente. Diferentes factores sanguíneos intervienen en la coagulación. La
hemofilia tipo A (la mas común) es aquella en la que falta el factor VIII.
Hasta recientemente el factor VIII se obtenía de donantes sanguíneos con el
consiguiente riesgo para la salud (SIDA, hepatitis...), actualmente se prepara
por biotecnología
a partir de bacterias que lo sintetizan merced a las técnicas de ADN
recombinante. La Reina Victoria de Inglaterra era portadora de la
enfermedad, el alelo fue pasado a dos de sus hijas y un hijo. Dado que las
familias reales de Europa generalmente se casaban entre sí, el alelo se
desparramó entre la realeza y pudo haber contribuido a la caída de los zares
rusos y a la llegada del comunismo al poder (el zarevich Alexei hijo del zar
Nicolás sufría de hemofilia A heredada de su madre, que portaba el gen secreto
de la reina Victoria, la influencia de Gregor Efimovic Novy, Rasputín "el
monje negro" en este reinado, era debida a que los zares pensaban que tenía
poderes curativos )
- La distrofia muscular es un término
que se aplica a una gran variedad de enfermedades del músculo. La mas
común es Distrofia Muscular de Duchenne (DMD), afecta los músculos
cardíaco y esqueléticos, como así también a algunas funciones mentales.
DMD es un factor recesivo ligado al X que tiene una frecuencia de 1 en
3500 nacimientos. Los que la sufren mueren generalmente antes de los 20
año. En 1987, Louis Kunkel informó el aislamiento de una proteína, la
distrofina, presente en individuos normales (cerca del 0,002 % de la
proteína muscular) pero ausente en dos individuos con DMD. La falta de
distrofina se acompaña con un proceso a nivel muscular conocido como
fibrosis, que restringe el suministro de sangre al músculo que por lo
tanto muere. La tecnología genética, se utilizó para secuenciar
y clonar
el gen de la distrofia, que es el mas grande que se conoce en humanos
(alrededor de 2 a 3 millones de pares de base), con 60 exones
e intrones
muy grandes.
- Una enfermedad genética de gran heterogeneidad: Con el nombre de retinosis pigmentaria se agrupa a una gran
cantidad de trastornos degenerativos de la retina causada por problemas en
los fotorreceptores, afecta a todos los grupos raciales en alrededor de 1
en 7000 personas, la "ceguera nocturna" y el "efecto túnel" son algunos de
sus síntomas. Se debe a la mutación de alguno de los numerosos genes que
codifican las proteínas de la fototransducción y dependiendo de cual puede
ser: autosómica dominante, recesiva o ligada al sexo.
Diagnóstico de las
enfermedades genéticas humanas
En la anemia drepanocítica se usaron para su estudio enzimas
de restricción, como la Hpa I. El ADN normal y patológico se sometió a la
enzima. Los fragmentos resultantes se sometieron a una sonda
radioactiva para el gen de la beta hemoglobina. La sonda hibridiza en la
hemoglobina normal con dos fragmentos de 7.000 a 7.600 nucleótidos de longitud.
En la anemia drepanocítica la hibridación ocurre con una sola secuencia de
13.000.
Un resultado similar se obtiene de los estudios de células obtenidas por
amniocentésis, lo cual provee una herramienta para la detección prenatal de la
anemia drepanocítica. Los marcadores
en los que ocurre la hibridación se conocen como RFLPs . El
fragmento grande en la anemia drepanocítica se interpreta como evidencia de una
mutación en la secuencia de reconocimiento. Dos secuencias nucleotídicas en la
misma molécula de ADN tienden a estar juntas (ligamiento).
En el ADN de la anemia drepanocítica la cadena beta de la hemoglobina quedo
ligado con otro gen que de alguna manera altera el reconocimiento de la
secuencia en la cual actúa la Hpa. Los heterocigotos tienen fragmentos largos y
cortos pero en los homocigotos dominantes y recesivos se encuentra un solo tipo
(corto o largo respectivamente) o sea:
- Homocigotos normales: fragmentos
cortos
- Homocigotos para la anemia
drepanocítica: fragmentos
largos
- Heterocigotos: fragmentos largos y
cortos
La enfermedad de Huntington fue estudiada
por James F. Gusella y sus colaboradores, que usó RFLPs (se pronuncia rif-lips)
para identificar un marcador. Realizando pruebas en una gran "librería"
de fragmentos de ADN humano, encontraron la aguja en el pajar. La enzima usada
fue la Hind III. Se identificaron cuatro fragmentos (A,B,C y D) en una familia
norteamericana cuyos miembros sufren la enfermedad. La presencia de un fragmento
A se identificó en individuos que sufren o sufrirán la enfermedad. El estudio
de
la genealogía de una familia venezolana de 3.000 miembros que desciende de un
marinero alemán que tenía la enfermedad de Huntington llevó a encontrar una
estrecha correlación entre el Fragmento C y la enfermedad. Muchos individuos no
desean saber si ellos desarrollaran la enfermedad.
La fibrosis quística también se estudió por la tecnología
RFLPs. Esta es la
enfermedad genética más común entre los caucásicos. Francis S. Collin ( director
del proyecto Genoma Humano desde 1993, entre sus logros se debe considerar su
participación en la identificación de los genes implicados en enfermedades
raras como la ataxia-telangestacia y la neoplasia endocrina múltiple tipo I ),
descubrió, en 1989 junto con sus colaboradores, el gen humano que al mutar
provoca la fibrosis quística.
Los
hemofílicos sufren de la falta de Factor VIII, hecho que puede detectarse en
fetos de 20 semanas. Un test mas preciso, que puede realizarse mas temprano, se
relaciona con el uso de una sonda
radioactiva (un fragmento de ARN de 36 nucleótidos) que hibridiza
fragmentos de restricción. El gen de la hemofilia tiene 186.000 pares de
bases, tiene 26 exones
separados por 25 intrones.
Las mutaciones en el gen pueden detectarse por RFLPs. Esta tecnología se ha
usado también para detectar la diferencia de un solo par de base entre la cadena
beta de la hemoglobina normal y la mutada.
Se ha desarrollado una sonda de ADN que hibridiza con el gen de la
distrofina. El test previo para la enfermedad de Duchenne, era para determinar
el sexo (con la opción de abortar si era macho). La nueva técnica permite
diferenciar entre machos sanos y enfermos, de esta manera los padres tienen mas
información para optar (si desean hacerlo). La hibridización solo ocurre si el
gen normal de la distrofina esta presente, no hay hibridación en los enfermos de
DMD.
El
genoma humano contiene secuencias de nucleótidos cortas e idénticas, alineadas y
repetidas miles de veces. Estas regiones difieren distintivamente de individuo a
individuo. Las mismas pueden escindirse del ADN original utilizando enzimas
de restricción, separarse por electroforésis
e identificarse por sondas
radioactivas. El método (ideado por A. Jeffreysen de la Univ. de Leicester,
Inglaterra) se conoce como "ADN fingerprint" ( "huellas digitales de ADN") dado
que al igual que las huellas digitales son únicas salvo el caso de gemelos.
El método es útil para establecer filiaciones e identificar sospechosos de
crímenes. Puede identificar el ADN en manchas de un vestido y conseguir que un
presidente de los EE.UU "reconozca que......, se arrepienta de........y.....bla...bla...bla.." y,
ufa!!!, ser responsable de la baja del Down-Jones y el Merval (la bolsa), todo por culpa del ADN fingerprint ( MALDITO
ADN FINGERPRINT, que linda frase para un afiche.....).
Combinado con el método de amplificación génica conocido como PCR (reacción
en cadena de la polimerasa; Mullis, K. B.; Nobel de Química 1993) que en pocas
horas puede obtener millones de copias de muy pocas moléculas de ADN, ha hecho
posible, entre otras cosas, obtener la "huella digital" de muy pequeñas
cantidades de sangre o semen, acelerar el diagnóstico prenatal de enfermedades
genéticas e identificar infecciones virales y bacterianas.
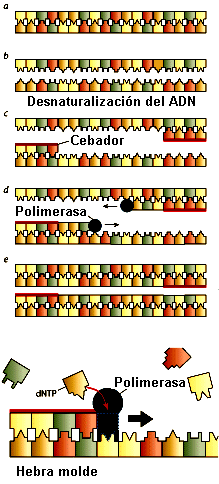
El método de la PCR implica
que en un tubo de ensayo se proceda a la separación de las cadenas de la doble
hélice por calentamiento, la adición de un cebador corto a una secuencia elegida
de ADN, la síntesis de la cadena complementaria por la acción de una ADN
polimerasa que trabaja a altas temperaturas (extraída de una bacteria "extremófila"), este ciclo se
repite unas 20 veces por lo cual el ADN presente se incrementa cerca de un millón de veces.
Trastornos genéticos "raros"

Existen
cerca de 5000 trastornos de origen genético clasificadas como "enfermedades
raras", por ejemplo el síndrome de Ehlers-Danlos (alteraciones del tejido
conjuntivo de las articulaciones), el síndrome de Marfan (tendencia a
aneurismas), el síndrome de Tourette (parpadeo, tos seca), porfiria intermitente
aguda y tantas otras. La Organización Nacional para el Control de Enfermedades
Raras de USA, tiene una base de datos de las mismas, de los especialistas y
tratamientos.(National Organization for Rare Disorder, Dept. RD, P.O. Box 8923,
New Fairfield, Conn. 06812) .


- Acondroplasia ( del griego a = privativo;
chrondos= cartílago; plassein = formar): Enfermedad caracterizada por la falta
del crecimiento o detención del desarrollo en longitud de los miembros.
- ADN complementario
(ADNc): ADN sintetizado a
partir de un molde de ARN; esta cadena no apareada suele utilizarse como sonda
(probe) en mapeos
físicos.
- Albinismo (del latín albus = blanco): El pigmento
melanina
no puede ser sintetizado por los albinos. Diferentes mutaciones pueden causar
albinismo: 1) la falta de una u otra enzima lo largo de la vía sintetizadora de
melanina; o 2) la incapacidad de la enzima para entrar en las células
pigmentarias y transformar el aminoácido tirosina en melanina.
- Alelo ( del griego allelon = el uno al otro,
recíprocamente): Formas alternativas de un gen, se hereda separadamente de cada
padre (p.ej. en el locus para el color de ojos puede haber un alelo para ojos
azules o uno para ojos negros. Uno o más de los estados alternativos de un gen.
- Amish (o
menonitas): comunidad religiosa no muy
grande, cuyos miembros se casan entre sí y, que por lo tanto tienen una
creciente incidencia de enfermedades genéticas. En Argentina existen colonias
menonitas en La Pampa y el Chaco. Los hombres casados se dejan la barba pero no
el bigote y las mujeres lleva vestidos largos y cofias. Entre sus
características apreciadas se encuentran su contracción al trabajo y otra, que
si fuera hereditaria habría que pensar como trasmitirla: pagan sus cuentas al
contado....
- Amplificación: Un aumento del número de
copias de un fragmento específico de ADN. Puede producirse in vivo o in vitro.
Ver clonación,
reacción en
cadena de la polimerasa.
- Amniocentésis: procedimiento invasivo por el
cual mediante una fina aguja que se inserta en el fluido amniótico (bajo
monitoreo ecográfico) que rodea al feto (término aplicado al bebé antes del
primer trimestre) se obtienen células que se desprendieron del mismo. Estas
células pueden cultivarse y utilizarse, entre otras, para determinar el
cariotipo el cual puede mostrar las anomalías del Síndrome de Down o el sexo del
bebé.
- ARN mensajero: "Molde" para la síntesis
proteica que es copiado de una de las hebras de ADN y que se traduce en los
ribosomas en una secuencia proteica. Se abrevia ARNm
- Autorradiografía (auto- forma prefijada
del griego autos = uno mismo): Impresión fotográfica que deja una sustancia
radioactiva cuando actúa sobre una película fotográfica sensible. Técnica que
utiliza film sensible a los Rayos X para visualizar moléculas marcadas o
fragmentos de moléculas marcadas; se utiliza para analizar el largo y el número
de fragmentos de ADN luego de su separación por electroforésis
en gel.
- Autosoma (auto- forma prefijada del griego autos
= uno mismo; soma = cuerpo): Cromosoma que no interviene en la determinación del
sexo. El genoma humano diploide consiste en 46 cromosomas compuesto de
22 pares de autosomas y un par de cromosomas sexuales (cromosomas X eY)
- Bases apareadas (en inglés base pair,
bp) Dos
bases nitrogenadas (adenina y timina o guanina y citosina) mantenidas juntas por
enlaces débiles (puente hidrógeno). Las dos hebras del ADN se mantienen juntas
formando una doble hélice por los enlaces de sus bases apareadas.
- Biotecnología: Aplicación práctica de los
avances de las técnicas utilizadas en la investigación básica de la bioquímica
del ADN, hoy utilizados en el desarrollo de productos por la industria del ADN
recombinante, fusión celular, manipulación genética de vegetales y otras.
- Braquidactilia (del griego brakhys = corto;
dactylos = dedos): Característica producida por un alelo de tipo dominante cuya
presencia determina que la segunda y tercera falange de los dedos sean más
cortas y por consiguiente los dedos son anormalmente cortos.
- Cariotipo (del griego karion = núcleo; typos =
estampar, imprimir): Microfotografía de los cromosomas individuales ordenados en
un formato normatizado que muestra el número, tamaño y forma de cada tipo de
cromosoma. Se usa para correlacionar anormalidades de cromosomas (observables a
este orden de resolución) con determinadas enfermedades.
- Cariotipo de flujo: uso de la citometría
de flujo para analizar y/o separar cromosomas en base a su contenido en ADN.
- Citometría de flujo: Análisis de material
biológico por medio de la detección de las propiedades de absorción de luz o de
fluorescencia de las células o fracciones subcelulares (p. ej. cromosomas) al
pasar por una estrecha abertura frente a una rayo láser. Se produce así un
perfil de absorbancia o fluorescencia de la muestra. Aparatos de fraccionamiento
automático de muestras, distribuyen las sucesivas gotas de la muestra analizada
de acuerdo a la fluorescencia emitida por cada gota.
- Cebador (en inglés primer): Cadena
polinucleotídica corta a la cual se agregan nuevos desoxirribonucleótidos por
acción de la ADN polimerasa.
- Centimorgan (cM, en honor de Morgan por sus trabajos
sobre genética): Unidad de medida de la frecuencia de recombinación. Un
centimorgan es igual al 1% de probabilidad que un marcador ubicado en un locus
se separe de otro marcador que se encuentra en un segundo locus por crossing-over en una generación. En seres humanos un centimorgan equivale en
promedio a un millón de pares de bases.
- Centrómero (del griego kentron = centro;
meros
= parte): región cromosómica (recubierta por el cinetocoro) donde se conectan
las fibras del huso durante la división celular.
- CEO
(del inglés Chief Executive Officer): El
"jefe" ejecutivo de un proyecto
- Clon (del griego klon = retoño): Grupo de células o
individuos originados de un solo progenitor, por reproducción asexual o por
manipulación biotecnológica. Uno de los miembros de dicho origen.
- Clonación: En la tecnología de ADN recombinante,
los procedimientos para la manipulación del ADN que permiten la producción de
múltiples copias de un gen o segmento de ADN se conocen como "clonación del
ADN". El proceso de producción asexual de un grupo de células u organismos
(clones), genéticamente idénticos.
- Código genético: Las secuencias de
nucleótidos (tripletes) que representan a los aminoácidos a introducir en una
molécula durante la síntesis proteica. La secuencia de nucleótidos del ADN puede
utilizarse para predecir la secuencia del mARN y por ende la secuencia de
aminoácidos.
- Codominante: caso en que, en los
heterocigotos, se expresan
ambos genotipos
presentes
- Colchicina: Alcaloide extraído de la semilla
del cólchico (Colchicum autumnale). Interfiere con las microfibrillas del huso
mitótico.
- Corea: movimientos
involuntarios incoordinados
- Cósmido: Vector de
clonación construido artificialmente que está flanqueado por las regiones
cohesivas (regiones COS) del bacteriófago
lambda. Los cósmidos pueden empaquetarse en el fago lambda e infectar
Escherichia coli, esto permite clonar
fragmentos mas grandes de ADN (hasta 45 kb) que los que se consiguen introducir
con plásmidos.
- Cromosomas(del griego chroma = color; soma =
cuerpo): Estructuras del núcleo de la célula eucariota
que consiste en moléculas de ADN (que
contienen los genes) y proteínas
(principalmente histonas).
- Crossing-over (del inglés entrecruzamiento):
Proceso que ocurre en la meiosis e incluye la ruptura de un cromosoma materno y
uno paterno(homologos), el intercambio de las correspondientes secciones de ADN
y su unión al otro cromosoma. Este proceso puede resultar en un intercambio de
alelos entre cromosomas.
- Deleción: la pérdida de un segmento cromosómico
sin que se altere el número de los mismos.
- Desórdenes poligénicos: resultantes de la
acción combinada de los alelos de más de un gen (p. ej. enfermedades cardíacas,
diabetes, ciertos tipos de cáncer). Si bien estos desórdenes son heredables,
dependen de la presencia simultanea de muchos alelos, por lo tanto
las pautas hereditarias son mucho mas difíciles que las relacionadas a un solo
gen.
- DOE: Department of
Energy, Departamento de
energía de los Estados Unidos
- Dominio: Porción de una proteína con una función
propia. La combinación de las funciones de los dominios de una proteína
determina su función global.
- Doble hélice: La forma que toman las dos
hebras de ADN cuando se encuentran unidas.
- Escherichia coli: Bacteria común,
habitante del intestino humano, estudiada intensivamente por los genetistas en
razón de su facilidad para crecer en el laboratorio, lo pequeño de su genoma y
su falta de patogenicidad (generalmente..)
- Electroforésis: Un método que permite
separar grandes moléculas (como proteínas o fragmentos de ADN) en una mezcla de
moléculas similares. Se pasa una corriente eléctrica por el medio de suspensión
y cada molécula. "viaja" por el medio a velocidades diferentes que dependen de
su tamaño y carga eléctrica.. La separación se basa en estas diferencias. La
agarosa y los geles de acrilamida se usan comúnmente para separar ácidos
nucleicos y proteínas.
- Endonucleasa (del griego
endon = dentro; asa
= sufijo que indica actividad enzimática): Enzima que corta una molécula de ADN
en un sitio interno de la secuencia de nucleotídica.
- Endonucleasa de restricción: Enzima que
reconoce específicamente determinadas secuencias y corta a la molécula de ADN en
ese sitio. De las bacterias se obtuvieron más de 400 enzimas que reconocen mas
de 150 diferentes secuencias de ADN (sitios de
corte por enzimas de restricción).
- Eucariotas (del griego eu = bueno, verdadero;
karyon = núcleo, nuez): organismos caracterizados por poseer células
con un núcleo verdadero rodeado por una membrana. El registro arqueológico muestra
su presencia en rocas de aproximadamente 1.200 a 1500 millones de años de
antigüedad
- Exón (del griego exo = por fuera):
La porción
del ADN de un gen que codifica para una proteína, los exones son característicos
de los eucariotas.
- Exonucleasa (xel griego exo = por fuera;
asa =
sufijo que indica actividad enzimática): Enzima que corta secuencialmente
nucleótidos de los extremos libres de un ácido nucleico
- Expresión génica: El proceso por el cual la
información codificada en los genes se convierte en las estructuras
operacionales presentes en las células. Los genes expresados incluyen a aquellos
que han sido transcriptos a ARNm y luego traducidos a proteínas y aquellos que
han sido transcriptos a ARN pero no traducidos a proteínas (p.ej. ARNt y ARNr)
- Expresividad y penetrancia: El grado
de expresión de un gen puede variar como resultado de su interacción con el
ambiente o con otros genes. Esta EXPRESION VARIABLE se ve en la polidactilia
(presencia de dedos supernumerarios en manos o pié), causada por una alelo
dominante. Existe una gran variabilidad en la expresión entre los integrantes de
la familia algunos tienen dedos supernumerarios en pies y manos y otros, solo en
el pié
Por otra parte se sabe que individuos que portan el gen dominante de la
polidactilia tienen manos y pies normales, es decir que el genotipo muestra una
penetración incompleta..
- Familia de Genes: Grupo de genes muy
relacionados que fabrican productos similares.
- Fitohemoaglutinina: producto de origen vegetal
(lectina) que aglutina eritrocitos, e induce a la mitosis.
- Gameto (del griego gamos = "unión de los sexos",
esposa): Célula reproductora haploide
(n) que cuando su núcleo se fusiona con otro gameto (n) del sexo opuesto
origina un cigoto (2n), que por mitosis desarrolla un individuo con células somáticas diploides (2n), en algunos hongos y protistas puede, por meiosis,
producir células somáticas haploides (n).
- Genes (del griego genos = nacimiento, raza; del latín genus =
raza, origen): segmentos específicos de ADN que controlan
las estructuras y funciones celulares; la unidad funcional de la herencia.
Secuencia de bases de ADN que usualmente codifican para una
secuencia polipeptídica de aminoácidos. Tema ampliado.
- Genoma: Todo el material genético de los cromosomas
de un organismo en particular, su tamaño se da generalmente como el numero total
de pares de bases.
- Genética: el estudio de la herencia de los
caracteres.
- Haploide (del griego haploos = simple, ploion =
nave): Célula que contiene solo un miembro de cada cromosoma
homólogo (número haploide = n). En la fecundación, dos gametos haploides se
fusionan para formar una sola célula con un número diploide (por oposición, 2n)
de cromosomas.
- Hemofilia (del griego haima, haimatos = sangre;
philos = amigo): desorden genético humano recesivo, ligado al sexo , resultante
de la ausencia de determinados factores que intervienen en la coagulación de la
sangre, generalmente Factor VIII. Los hemofílicos tienen dificultades en la
coagulación de su sangre.
- Hibridación (del latín ibrida = "producto de
la cruza de dos animales diferentes"): El proceso de "juntar" dos hebras
complementarias de ADN o una de ADN y otra de ARN para formar una molécula
bicatenaria.
- Hibridación "in situ" (del latín "en el
lugar"): El uso de sondas de
ADN o ARN para detectar la presencia de secuencias de ADN complementarias a
ellas, en bacterias clonadas o células eucariotas cultivadas.
- Heterocigoto (del griego heteros = diferente)
La presencia de alelos
diferentes en el mismo locus de
cromosomas homólogos. Cuando los dos alelos son diferentes, el alelo dominante
es el que se expresa.
- Hipotónico (del hypo = debajo; tonos = tensión:
De dos soluciones, la de menor concentración de solutos. Si se encuentran
separadas por una membrana que no deje pasar el soluto (semipermeable) el agua
se mueve desde la de menor concentración hacia la de mayor concentración.
- "Homebox" genes: Corta secuencia de
nucleótidos virtualmente idéntica en todos los genes de los organismos que la
contienen. Se la ha encontrado en numeroso organismos, desde la mosca de la
fruta a los seres humanos. En la mosca de la fruta parecen determinar los grupos
particulares de genes que se expresan durante el desarrollo.
- Homocigoto (del griego homos = mismo o similar,
zygon = par): Cuando los dos alelos son
iguales.
- Homología ( del griego homologia = acuerdo):
similitudes entre las secuencias de ADN o proteínas entre individuos de la
misma especie o entre especies diferentes.
- Human Genome
Sciences: companía especializada en encontrar genes que codifican
proteínas solo en base a ADNc. HGS pretende utilizar como fármacos los
genes y las proteínas que éstos producen y ya ha conseguido
aprobación de la FDA para ensayar
cuatro fármacos de origen genético entre ellos el estimulador del
linfocito B.
- Informática: El estudio de la aplicación de la
computadora y técnicas estadísticas al manejo de la información. En el proyecto
genoma la informática desarrolla métodos para la búsqueda rápida en bases de
datos, analizar la información proveniente de las secuencias de ADN y para
predecir la secuencia de las proteínas a partir de la secuencia de ADN.
- Intrón: La secuencia de bases de ADN que interrumpe
la secuencia de un gen que codifica para una proteína, esta secuencia se
transcribe al mARN pero en un proceso de "corte y empalme" se separa del mismo
antes que el mARN sea traducido a proteína.
- "in vitro": del latín literalmente "en
vidrio", se usa para indicar experimentos realizados fuera de un organismo vivo
- Kilobase (kb): Unidad de longitud del ADN
equivalente a 1000 (1k) nucleótidos.
- Librería: una colección desordenada de clones
(por ejemplo ADN clonado de un organismo particular) cuyas interrelaciones pueden
establecerse por mapeo físico
- Ligamiento (del inglés
linkage): La proximidad
de dos o más (genes) marcadores en un cromosoma; cuanto mas próximos se
encuentran los marcadores menor es la posibilidad que se separen durante los
procesos de división celular y por lo tanto mayor la posibilidad de que se
hereden juntos.
- Locus (pl. loci, del latín lugar): Posición de un
gen en un cromosoma.
- Mapeo genético: Determinación de la
posición relativa de los genes en la molécula de ADN (plásmido o cromosoma) y
las distancias (unidades de "unión") entre ellos
- Mapa comosómico: Diagrama que muestra las
posiciones relativas en un cromosoma de los loci de los
genes, basado en la frecuencia en que heredan conjuntamente. Las distancias se
miden en centimorgans (cM).
- Marcador (del inglés marker; pob. del italiano
marcare = "señalar una cosa para que se distinga de otra"): Una posición
física identificable en un cromosoma cuya herencia puede seguirse (p.ej. un gen
o un sitio que corta una enzima de restricción). Los marcadores pueden ser una
región del ADN que se expresa (gen) o un segmento de de ADN que no se conoce que
codifica pero que se puede seguir su manera de heredarse( ver RFLP iniciales del
inglés Restriction Fragment Lenght Polymorphism)
- Melanina (del griego melan= negro): pigmento que
da color a la piel y protege a la capas subyacentes de la radiación ultravioleta
- Megabase (Mb): Unidad de longitud del ADN
equivalente a 1.000.000 de nucleótidos aproximadamente igual a 1 cM.
- Meiosis (del griego meio = menor): División
celular en la cual la copia de los cromosomas
es seguida por dos divisiones nucleares. Cada uno de los gametos resultantes
recibe la mitad del número de cromosomas número (número haploide) de la célula
original.
- Mapa Físico: mapa de la localización de marcas
identificables en el ADN (p. ej. genes, sitios de corte de enzimas de
restricción, bandas) que se confecciona prescindiendo de los fenómenos
hereditarios. Las distancias se miden en pares de bases. En el genoma humano el
mapa físico de de menor resolución lo constituye los patrones de bandeo de los
24 cromosomas diferentes (22 autosómicos, el X y el Y). El de mayor resolución
lo constituirá la secuencia completa de nucleótidos de los cromosomas.
- Moleculas de ADN recombinante:
Combinación de moléculas de ADN de diferentes orígenes que se unen por medio de
la tecnología del ADN recombinante.
- NIH: (National Institute of Health) Instituto
Nacional de la Salud de los Estados Unidos
- Oncogén (del griego onkos = tumor): gen asociado
al desarrollo del cáncer. Muchos oncogenes están directa o indirectamente
relacionados con el control de la división celular.
- Polimerasa (ADN o ARN): Enzimas que catalizan
la síntesis de ácidos nucléicos en base a templados preexistentes, utilizando
ribonucleótidos para el ARN y desoxirribonucleótidos para el ARN.
- Polimorfismo: Diferencia entre las secuencia
de ADN entre individuos.
- Promotor: Sitio del ADN donde se pegará la ARN
polimerasa para iniciar la transcripción.
- Producto génico: El material
bioquímico, ya sea ARN o proteína, resultante de la expresión de los genes. La
cantidad de producto génico se utiliza para medir cuan activo es un gen,
cantidades anormales pueden correlacionarse con alelos que causan enfermedades.
- Proyecto Genoma Humano: Nombre que
abarca una serie de proyectos iniciados en 1986 por la DOE y
apoyados, entre otros, por la NIH para 1) crear un registro
de segmentos de ADN de localizaciones cromosómicas conocidas 2) Desarrollar métodos informáticos
para analizar el mapa genético y los datos de las secuencias de ADN y 3)
Desarrollar nuevas técnicas e instrumentos para analizar y detectar ADN. Tema
ampliado
- Reacción en cadena de la
polimerasa (PCR, de las iniciales en inglés Polimerase Chain
Reaction):
Método de amplificación
de una secuencia de bases del ADN usando una polimerasa termoestable y dos
cebadores ("primers")
de 20 bases de largo de la secuencia a ser amplificada, uno complementario de
las secuencias final de la hebra (+) y otro de la otra secuencia final de la
hebra (-). En razón que las nuevas cadenas de ADN sintetizadas pueden
subsecuentemente servir de moldes adicionales para la misma secuencia de
cebadores, sucesivos "ciclos" de anillado de cebadores, alargamiento de la
cadena y disociación del ADN bicatenario formado producen rápidamente grandes
cantidades de la secuencia original (amplificación). La PCR puede utilizarse
para detectar una secuencia definida en una muestra de ADN.
- Recesivo: Término que se aplica
a un carácter (alelo) que solo se expresa
cuando el segundo carácter (alelo) es igual.
- Recombinación: El proceso por el cual se produce en
la progenie una combinación de genes diferentes a los de los padres. En los
organismos superiores por el proceso de entrecruzamiento (crossing over).
- Replicación del ADN ( del latín
replere = rellenar): El uso de un ADN existente como molde para la síntesis de
nuevas hebras de ADN. Proceso que en eucariotas ocurre en el núcleo.
- RFLP: (del inglés restriction-fragment-length
polymorphisms; polimorfismo de longitud de los fragmentos de restricción):
Bajo esta sigla se reconoce las variaciones entre individuos de la longitud de
los fragmentos de ADN cortados por enzimas de restricción. Este hecho
generalmente es causado por mutaciones en el sitio de corte.
- Secuenciar: Determinación del orden de los
nucleótidos en el ADN o ARN o el orden de los aminoácidos en las proteínas.
- Secuencia de bases: el orden de las bases
de los nucleótidos en una molécula de ADN.
- Secuencia complementaria: Secuencia
de bases en una molécula de ADN que puede formar una doble hebra al aparear las
bases de otra. Por ejemplo la secuencia complementaria de G-T-A-C es
C-A-T-G.
- Secuencia conservada: Secuencia
de bases en una molécula de ADN (o de aminoácidos en una proteína) que ha
permanecido prácticamente intacta a lo largo de la evolución.
- Síndrome de Down: Aquellos con
el síndrome de Down sufren de retardo mental moderado o severo, cuerpo bajo,
macizo, cuello grueso y lengua agrandada lo que dificulta la dicción. Son
propensos a infecciones y enfermedades cardíacas y (en aquellos de mayor
supervivencia), a desarrollar la enfermedad de Alzheimer. El 95% de los casos
de Down son el resultados de la no disyunción del cromosoma 21.
- Sitios de corte por enzimas de restricción: Una
secuencia de bases específica en la molécula de ADN al cual corta una particular
enzima de restricción. Algunos sitios se encuentran frecuentemente en el ADN
(por ejemplo cada cientos de pares de bases) otro son mucho menos frecuentes por
ejemplo cada 10.000 pares de bases.
- Sonda (del francés sonde , abreviación del
anglosajón sundline, en su acepción de elemento exploratorio): Molécula de ADN o
ARN monocatenario con una secuencia de bases específica, marcada radiactivamente
o inmunológicamente, que se utiliza para detectar secuencias de
bases complementarias por hibridación
- Terapia génica (del griego therapeuo = "yo
cuido"): Inserción de ADN normal en una célula para corregir un defecto
genético.
- Tecnología del ADN recombinante:
Procedimientos utilizados para unir segmentos de ADN en un sistema libre de
células ("cell free system" es decir fuera de un organismo o célula). Bajo
condiciones adecuadas, una molécula de ADN recombinante puede introducirse
dentro de una célula para ser replicado, ya sea autónomamente o integrada al
cromosoma celular.
- Translocación: fenómeno por el cual un
fragmento de un cromosoma se transfiere a un cromosoma no homólogo.
- Transferencia horizontal de
genes: mecanismo por el cual se transmiten genes individuales, o grupos de ellos, de una especie a otra
- Tumor de Wilm: Cáncer de riñón en niños y bebés
asociado a una deleción
del cromosoma 11. Asociada con esa deleción existe una efermedad denominada
aniridia (ausencia del iris en el ojo). Cuando hay aniridia y deleción existe
gran riesgo de desarrollo del tumor.
- Vector de Clonación: (del latín vehere =
transportar) Molécula de ADN originada en un virus, plásmido, o en la célula de
un organismo superior en el que se puede integrar otro fragmento de ADN, sin que
pierda la capacidad de autoreplicación de. Los "vectores" introducen ADN extraño
en una célula huésped, donde puede reproducirse en grandes cantidades. Ejemplos:
plásmidos, cósmidos y los cromosomas artificiales de levadura. A menudo los
vectores son moléculas de ADN recombinante que contienen secuencias de
diferentes vectores.

Traducción, redacción y diagramación a cargo de :
Dr. Jorge S. Raisman, lito@unne.edu.ar
Ing. Ana María
Gonzalez, amgonza@unne.edu.ar Actualizado
en Junio de 2000. Reproducción autorizada únicamente con fines educativos citando su origen.
Se agradecen comentarios y sugerencias.
|


![]()