RhadbovirusSteve Dewhurst, Associate Professor of Microbiology and
Immunology, University of Rochester Medical Center USA.
|
|
|
Características comunes de los mononegavirus:
- Genoma: linear monopartido (-) ARN
- Organización del genome: 3'-[SIN TRADUCIR. líder]-[NUCLEO]-[ENVOLTURA]-[POL]-[SIN TRADUCIR]-5'
- Virión: nucleocápside en espiral ó el núcleo de la ribonucleoproteína (RNP) contiene una polimerasa ARN dependiente del ARN viral y otras proteínas. El ARN viral por sí mismo no es infeccioso.
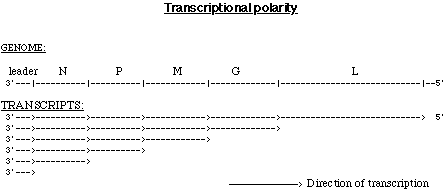
- Transcripción: La transcripción del ARNms se hace por síntesis programada con señales de parada y arranque a partir de un único promotor. Señales transcripcionales discretas delinean los genes.
- Replicación: la replicación del ARN ocurre cuando una secuencia completa de ARN(+) se ha hecho. Esta secuencia actúa a su vez como modelo para la fabricación de los demás genomas. La replicación requiere nuevas síntesis proteicas y solo pueden sintetizarse si las señales de parada y arranque son ignoradas.
Esquema genérico de la replicación de los mononegavirus

Filoviridae
Historia, epidemias:
En 1967 aparecieron brotes simultáneos de fiebres hemorrágicas en Yugoeslavia y Alemania, en trabajadores de laboratorio que estaban procesando riñones procedentes de los monos verdes africanos. Hubo 31 casos y 7 fallecidos. El virus se caracterizó por primera vez en Marburg, Alemania y se averiguó que provenía de un único cargamento de monos de Uganda. Casos esporádicos han ocurrido después en 1975, 1980, 1982 y en 1987.
Zaire y Sudán padecieron en 1976 epidemias severas de fiebre hemorrágica, con 550 casos y 430 muertes. Se aisló el virus de Ebola (nombre recibido de un río en el NW del Zaire). La ultraestructura del virus se parece al virus de Marburg, pero es antigénicamente (y genéticamente) distinto. Ahora parece que existen al menos 3 y probablemente 4 virus EBO: EBO-Z (Zaire), EBO-S (Sudán), EBO-IC (Ivory Coast = Costa de Marfil) y EBO-R (Reston, USA). Los dos primeros son muy letales en los humanos y se propagan por medio de los fluídos corporales por contacto (no sexual) habitual con los animales. El virus Reston, parece que es menos letal en los humanos (6 casos, 0 muertes), aunque es letal en los primates no humanos.
Los brotes epidémicos de Ebola ocurrieron en 1995 en el área de Kikwit del Zaire (315 casos y 244 muertes) y en el NW de Gabón, la zona de Mayibout (37 casos y 21 muertes). No está claro cómo empezó éste último brote. El brote de Gabón empezó en un grupo de 20 jóvenes de Gabón que atraparon un chipamcé enfermo. Se cree que los jóvenes enfermaron en la preparación del chipamcé para cocinarlo. El virus de Ebola se ha aislado solamente de los chipamcés comedores de carne y no de los chipamcés estrictamente vegetarianos pertenecientes al mismo grupo animal.
Los brotes de EBO-Reston ocurrieron en las colonias de primates del área de Washington (Reston, 1989) y en Texas (1990, 1996). estos brotes fueron contenidos con la destrucción de los animales dentro de la zona afectada donde estaban. Al parecer el virus vino en los cargamentos de macacos que venían de un único exportador filipino. Ninguno de los 6 humanos infectados con el EBO-R ha muerto de la enfermedad.
Finalmente, la ruta principal de transmisión del Ebola es el contacto con los fluídos corporales infectados y con la sangre infectada (lo que puede ocurrir en el personal sanitario, personal de funerarias, etc) es posible que algunos virus Ebola puedan transmitirse por vía respiratoria, por medio de los aerosoles de un estornudo,ect. Este supuesto se basa en la evidencia de que los monos rhesus que convivían en la misma habitación, pero que no tenían contacto directo con monos inoculados de modo experiemntal con el EBO-Z, se infectaron (Jaax et al. Lancet 346:1669, 1995).
Los filovirus producen las infecciones clásicas emergentes: Los filovirus son agentes de seguridad biológica de nivel 4 (el HIV es de nivel 2+ solamente). Son filamentosos con un genoma linear de ~13-19kb. Pueden infectar a los ratones, hamsters, conejillos de indias y a los monos, y no se conoce el reservorio viral en la naturaleza. Las epidemias humanas parecen estar relacionadas por el contacto con sangre infectada en los nosocomios. (a veces por la reutilización de agujas en los hospitales = nosocomios) y por contacto con las personas infectadas. Las infecciones primarias con el Marburg y Ebola fueron 25-90% fatales. Se cree que la muerte se debe a la necrosis de los órganos viscerales (como el hígado), producido por la infección viral. No está claro el papel de la hemorragia en la muerte de la persona afectada. Los monos salvajes traídos a USA deben pasar una cuarentena antes de ser llevados a los centros de primates.
Los virus clonados ayudarán al desarrollo de los test serológicos para el diagnóstico de la infección., y se progresa en el desarrollo de una vacuna para el virus de Ebola. La primera vacunación con éxito en contra de este virus se hizo en 1998 por el grupo de Gary Nabel de la Universidad de Míchigan. En el informe, la vacuna ADN que codifica la glicoproteína del virus de Ebola fué capaz de inducir una respuesta inmune basada en las células T en los conejillos de Indias, suficientemente potente para proteger a los conejillos de la infección por el virus vivo de Ebola (Xu et al. Nature Medicine 4:37, 1998).
Genética de los Filovirus. Los análisis de las secuencias de los virus de Ebola, en los brotes de 1976 y 1995, han revelado un sorprendente alto nivel de conservación genética para un virus ARN. Una interpretación de la estabilidad de estas secuencias, es que los virus Ebola han co-evolucionado con sus reservorios naturales y no han cambiado substancialmente en el ambiente silvestre. (vea más abajo).
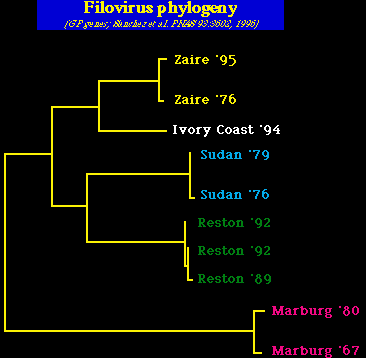
En líneas generales, los filoviridae están más relacionados con los paramyxovirus que con los rhadbovirus. Los genomas han sido secuenciados en su totalidad y se han identificado dos gruppos principales: Marburg y Ebola.
Las propiedades únicas de los filovirus, comparados con otros mononegavirus son:
1- Tienen el tamaño del genoma más grande.
2- La secuencia 3'- UAAUU se encuentra entre la mayoría de los genes
3- Hay varios genes que se solapan
4- Dominio putativo inmunosupresivo en los productos genéticos GP
Los virus de Margurg y Ebola difieren en que el Marburg GP es codificado en una secuencia única de lectura abierta, mientras que el Ebola GP es codificado en dos secuencias de lectura. La expresión del Ebola GP, necesita de un lugar ARN específico de edición, análogo a lo que pasa en el virus del sarampión. Específicamente, un residuo A no secuenciado por una plantilla se añade al ARNm, lo que permite que se unan las dos secuencias de lectura abiertas.
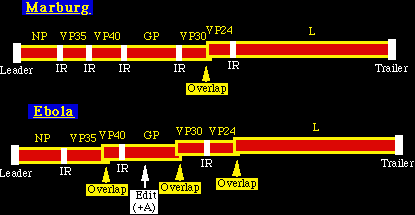
Estructuras de los genomas
de los Filovirus
IR: intervening regions (regiones que intervienen); GP: viral glycoprotein
(glicoproteínas virales); VPxx: viral proteins (proteínas virales); Editing site (siio
de edición) : addition of a nontemplated A ( adición de un fragmento A no
secuenciado de una plantilla)
Virus de la enfermedad de BoARN
Patogénesis: El virus de la enfermedad de BoARN (BDV) es un agente neurotrópico que infecta a los caballos y a las ovejas, y es capaz de infectar a los primates. No se ha encontrado en los humanos. La enfermedad inducida por el BDV reproduce una enfermedad mental parecida a la ezquizofrenia. La replicación viral no es citolítica y por eso hay pocos virus libres liberados a la circulación.
Genética: El genoma completo del BDV ha sido secuenciado se han identificado 3 bloques de secuencias conservadas, similares a las encontradas en los mononegavirus. De izquierda a derecha son:
1- Cofactores de la nucleoproteína y de la polimerasa
2- Matrix y envoltura
3- Polimerasa viral
Aspectos únicos moleculares del BDV son:
1- Existe una replicación y transcripción nuclear, con un nivel alto de ARNms ramificado
2- El BDV está relacionado con el genus de los virus de las plantas: Nucleorhadbovirus
Rhadboviridae
Del griego "rhadbo": en forma de palo, ó de bastón
Existen más de 100 rhabdovirus. Infectan a casi todos los animales. Dos géneros infectan a los mamíferos:
- Los Lyssavirus: invaden el CNS; (fr. Griego "lyssa": frenético) ejemplo: virus de la rabia.
- Vesiculovirus: invaden las células epiteliales (la lengua) y producen vesículas. Ejemplo: el virus de la estomatitis vesicular (VSV).
Rabia: Causa una encefalitis en los animales y humanos infectados por mordeduras, pero algunos animales son más susceptible que otros (las zorras y lobos > perros, mofetas, mapaches > opposum.) Se propaga a los humanos por mordeduras de animales afectados. En USA es más prevalente en las mofetas, pero también se encuentra en los mapaches y algunas veces en los murciélagos. En las demás partes, es más frecuente en los humanos debido a su presencia en los perros.
Rabia de los murciélagos: En los últimos 20 años, han ocurrido menos de 30 casos de rabia indígena en USA. En la mayoría de los casos, las mordeduras no fueron documentadas y, en casi la mitad de los casos, los análisis secuenciales revelaron que la infección se debía a un virus de la rabia procedente de los murciélagos. La mayoría son variantes que infectan a los murciélagos de pelos plateados (SHBRV = silver-haired bats rhabdo virus ). estos murciélagos pertenecen a una especie relativamente rara y no se asocian generalmente con humanos, y forman una minoría dentro de todos los murciélagos con la rabia de USA (En general, menos del 1% de los murciélagos dan (+) el test de la rabia y los murciélagos que se encuentran con más frecuencia entre los humanos, los murciélagos marrones, no se ha demostrado nunca que produzcan enfermedad en los humanos). La rareza de los murciélagos de pelos plateados y su más rara asociación con los humanos sugieren que algo inusual está pasando. Hay más datos que muestran que las variantes del SHBRV se replican con una eficiencia muy alta en los cultivos celulares de las células epiteliales, especialmente a bajas temperaturas (34ºC). Lo que permite que el virus se reproduzca mejor en la piel. (Morimoto et al. PNAS 93:5653, 1996).
Cuadro clínico: La enfermedad varía según la localización y severidad de las mordeduras (típicamente ~15% de infección). El inicio de la enfermedad es lento, con un período largo de incubación (puede ser > un año), durante el cual el virus se reproduce en el músculo más cercano a la mordedura. Después el virus penetra en los nervios periféricos y de aquí viaja a los ganglios basales y entra al cerebro. Entonces se disemina a todos los tejidos (incluyendo las glándulas salivales). La muerte es inevitable si el virus entra a los nervios, por eso la intervención médica después de la mordedura es exitosa casi siempre.
Control de la rabia: se consigue controlando el reservorio animal, ya por vacunación de los animales domésticos como por el uso de cebos con vacuna para los animales salvajes.
Tropismo: El virus de la rabia es neurotrópico. Entra los nervios usando parcialmente el receptor de acetilcolina, pero el virus puede infectar también a las células AChR(-), lo que sugiere que la penetración en el nervio utiliza otros receptores. In vitro, el virus de la rabia es moderadamente citopático.
VSV: causa epidemias, pero es una enfermedad que se limita al ganado. Puede infectar a los cerdos, caballos, humanos y aún a los insectos (tiene un espectro amplio de huéspedes). En los humanos produce una enfermedad parecida al resfriado y es bastante común en los trabajadores de los laboratorios. El VSV receptor no es una proteína (la tripsinación prolongada de los cultivos celulares no bloquea la infección). Puede ser la fosfatidil serina.
Citopatogenecidad. Lo induce rápidamente in vitro. El virus puede ser estudiado convenientemente por análisis de placa ( exposición de una monocapa de células cultivadas al VSV, se lava y se cubre con un medio semi-sólido, lo que permite la difusión del virus a las células vecinas, pero previene la contaminación por convección a otros lugares de la placa).
Biología molecular / Genomas del Rhabdovirus
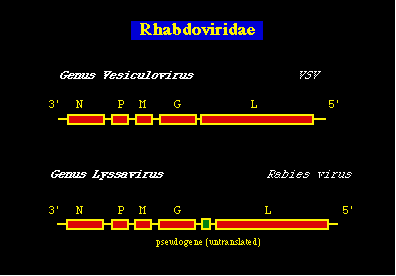
RABIA: El genoma del virus de la rabia es muy similar
al VSV. La mayor diferencia es que las regiones intergénicas son más largas y más
divergentes en el virus de la rabia, el virus también contiene un pseudogene no
traducido. La proteína más estudiada del virus de la rabia es la proteína G, es muy
importante para la vacunación, ya que es el blanco de los anticuerpos neutralizantes.
También es importante para la patogénesis, ya que las cepas atenuadas de la rabia
contienen una mutación en el residuo del aminoácido 333 en comparación con las cepas
salvajes. Este hecho da lugar a la pérdidad del poder patógeno, una disminución en la
cinética de su propagación dentro del SNC y una menor efiiciencia de infectar a las
células nerviosas, in vitro.
Biología molecular del VSV
En general, la biología molecular del VSV está mucho más entendida que la del virus
de la rabia
La morfología y estructura del VSV es similar al virus de la rabia. Las partículas tiene forma de balas y se compone de dos estructuras principales: la nucleocápside ó ribonucleoproteína (RNP) del núcleo y una lipoproteína que rodea el núcleo a modo de envoltura.
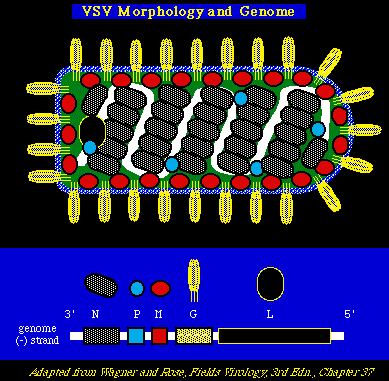
Estructura de las proteínas virales
NUCLEO RNP VIRAL: La nucleocápside ó RNP ( ribonucleoproteína) del núcleo es el componente infeccioso del VSV y de todos los demás rhabdovirus. Tal y como se muestra en el diagrama, el núcleo incluye el genoma ARN viral, el cual está fuertemente asociado con la abundantísima proteína N de la nucleocápside. El núcleo del RNP contiene además otras proteínas en menor cantidad, la fosfoproteína (P) y la polimerasa ARN viral (L). La abundancia relativa de éstas proteínas, por virión es como sigue: approx. 1250 moléculas de N, 500 moléculas de P y solamente 50 moléculas de L. (No es sorprendente, ya que L es una enzyma y las otras son por naturaleza proteínas estructurales).
Protein N: La función de la proteína N parece ser:
1- promover la encapsidación ó enpaquetamiento del ARN
2- permitir la replicación del genoma, favoreciendo la transcrpción por el mecanismo de
permitir que la polimerasa viral lea por encima de las señales de parada y/o arranque,
localizadas entre los genes virales.
Proteina P: La función de la proteína P, que es muy acídica debido a la fosforilación, parece actuar:
1- como un cofactor de la polimerasa, posiblemente ayudando a desplazar la proteína N
del ARN viral y
2- unirse a la proteína N, tal vez permitiéndola que encapside al ARN viral (un paso
importante en el empaquetamiento del ARN).
Proteina L: Es el ARN viral controlado por la polimerasa ARN. No es activa por sí misma, ya que la proteína P se necesita para la actividad catalítica.
ARN viral : Tal y como se muestra en el diagrama, el genoma VSV contiene 5 genes en el orden 3' N-P-M-G-L 5'.. El gen P también codifica dos productos más pequeños desde una segunda secuencia de lectura, aunque sus funciones no se conocen bien. cada uno de estos genes está separado del otro por una secuencia muy corta de solo 2 nucleótidos.
Envoltura viral Los mayores componentes de la envoltura del VSV son:
1- la glicoproteína viral (G) anclada a
la membrana
2- la mattrix (M). Se encuentran cantidades,
más ó menos equivalentes, de las dos proteínas en el virión. (apprx. 1500 moléculas
por virión).
Proteina G: La glicoproteína G, forma puntas triméricas en la superficie de la partícula viral y forma en el virus, tanto los principales determinantes antigénicos como las principales moléculas capaces de unirse al receptor. La proteína G sufre un cambio de adaptación a un pH levemente ácido (pH < 6.0), lo que le permite estabilizar al trímero y exponer un dominio hidrofóbico que puede insertar en la membrana celular y dar lugar a la fusión con la membrana. Así, la VSV fusión es activada en la vesícula endocelular, en respuesta al cambio ácido del pH.
Proteina M: Es una proteína muy basófila, se cree :
1- juega un papel en el ensamblaje viral
2- participa en la inhibición de la síntesis del ARN viral para permitir que se inicie
el enpaquetamiento (puede ser como consecuencia de la condensación del núcleo RNP).
Expresión del gen viral
Después de su entrada en la célula huésped y de que se haya decapado(queda el núcleo desnudo) el núcleo RNP, el VSV empieza a expresar sus genes. Como el genoma viral es de sentido negativo (tiene una polaridad opuesta al ARNm), el primer paso es la transcripción del ARNms viral. NOTA: el núcleo RNP es activo en su capacidad de transcribir, y no es inhibido por la actinomicina-D (a diferencia de las polimerasas ARN celulares, que usan una plantilla ADN para dirigir la síntesis del ARNm).
La transcripción viral se inicia en el 3' del final del genoma viral, en un único promotor, y continúa secuencialmente a loa largo del genoma. Se cree que el ARNm de longitud = unidad génica se producen por mecanismos de transcripción con parada/arranque (ver más abajo). Uno de los resultados de este proceso es de que la transcriptasa sufre pausas y la transcripción es atenuada en un 30% en cada unión de genes. Lo que a su vez produce un gradiente de producción de ARNm , como N>P>M>G>L.
La transcripción parada/arranque, se consigue por la presencia de señales de transcripción en las fronteras de los genes. Hay una señal de inicio en 5' y tambien en 3'-poliA y señales de finalización, las cuales están ordenadas: [polyA-señal/terminar]--[región intergénica]--[iniciador]. Nota: la región intergénica (2 nucleótidos) no se transcribe durante la síntesis del ARNm
Replicación del ARN viral
A diferencia de la transcripción del ARNm viral, la replicación del ARN viral requiere que el virus forme una copia única completa de su genoma. Así, la diferencia en la replicación está en que las señales de parada/arranque transcripcionales deben ser ignoradas de algun modo
La decisión para replicar el genoma viral debe ser hecha cuando se encuentra la primera región (localizada entre la región que codifica la secuencia sin traducir, corta del lider del ARN, y el gen que codifica la proteína N). Esta región intergénica debe ser leída en su totalidad, para que pueda ocurrir la replicación del ARN viral.
Es interesante que, la replicación del ARN viral requiere una traducción activa (ha sido probado de modo experimental, ya que la replicación ARN viral, pero no la síntesis de ARNm viral, se bloqueó inhibiendo la síntesis de proteínas usando la cicloheximida). Esta observación es consistente con un modelo en el cual la proteína N recién formada, se une selectivamente a la secuencia lider ARN viral. Haciéndolo así, la proteína N evita que se reconozcan las señales transcripcionales de finalización. De este modo el cambio del síntesis de ARNm a la replicación del ARN se regula principalmente por la actividad anti-terminación de la proteína N.
Nota: la longitud total del ARN viral en sentido (+) no contiene ninguna secuencia regulatoria de la transcripción, y por lo tanto funciona solamente como una plantilla para la producción de una cadena de longitud completa de sentido (-) (como es la producción de nuevos genomas virales).
Genética molecular y vectores
La replicación del VSV ha sido muy difícil de estudiar usando los métodos del ADN recombinante. Se debe a que el ARN desproteinizado, no es infeccioso. De igual modo, el ARN transcrito de clones cADN, no es por sí mismo capaz de iniciar la infección. La polimerasa ARN viral es necesaria para iniciar la infección y además el ARN debe estar encapsidado para servir de plantilla funcional para la polimerasa. Finalmente,es necesaria la síntesis constante de proteínas N para la replicación.
Recientemente, se ha hecho un gran progreso en esta área. Se han desarrollado métodos para producir virus infecciosos de VSV y del virus de la rabia a partir de clones del cADN. Esto permitirá análisis inversos genéticos de las funciones genéticas del VSV y del virus de la rabia por mutaciones dirigidas a un sitio específico. Además, se han producido vectores basados tanto en el VSV como en el virus de la rabia, dichos vectores tienen un espectro amplio de huéspedes y pueden ser muy útiles.
Clones de Rhabdovirus cADN infecciosos
Schell et al. EMBO J. 13:4195, 1994; Lawson et al. PNAS 92:4477, 1995; Whelan et al. PNAS 92:8388, 1995; Mebatson et al. PNAS 93:7310, 1996
Explicacion: los círculos vede oscuros representan las monocapas celulares y las
manchas blancas son las placas producidas por el virus infeccioso. Puede verse que el
virus infeccioso fué producido SOLAMENTE cuando una copia intacta del cADN del genoma del
VSV fué introducida en las células junto con los plásmidos de soporte que codifican las
proteínas (N, P, y L) que componen al RNP viral. La última placa, demuestra que
las manchas blancas se debían a la presencia del VSV, pues la formación de la mancha
blanca fué bloqueada por la adición de suero con anticuerpos neutralizantes en
contra del VSV (anti-VSV Ab).
Actualizado 1/99, Steve Dewhurst, Associate Professor of Microbiology
and Immunology
© Copyright University of Rochester and Stephen Dewhurst, 1996-99
URL: http://www.urmc.rochester.edu/smd/mbi/grad2/nns99A.html